Modern technologies for studying the genome of mycobacteria
DOI:
https://doi.org/10.15587/2519-4798.2023.290596Keywords:
Amplification, Genome, Hybridization, Molecular Diagnosis, Mycobacterium, Restriction, Sequencing, Spoligotyping, TuberculosisAbstract
Molecular technologies play a leading role in the laboratory diagnosis of tuberculosis and mycobacteriosis. The successes in studying the genome of Mycobacterium have contributed to significant progress in understanding the evolution, variability, and genetic diversity of pathogens, as well as the development of diagnostic technologies, including research into resistance to anti-tuberculosis drugs.
The aim of this research is to conduct a comparative study of the spectrum of modern technologies for studying the genomes of mycobacteria and their impact on the efficiency of the laboratory diagnosis of tuberculosis.
Materials and methods: a search for sources of information was carried out in the PubMed, Medline, Web of Science, and Google Scholar databases. Materials related to the technology of molecular diagnosis of tuberculosis and mycobacteriosis and for determining the susceptibility of pathogens to anti-tuberculosis drugs were selected.
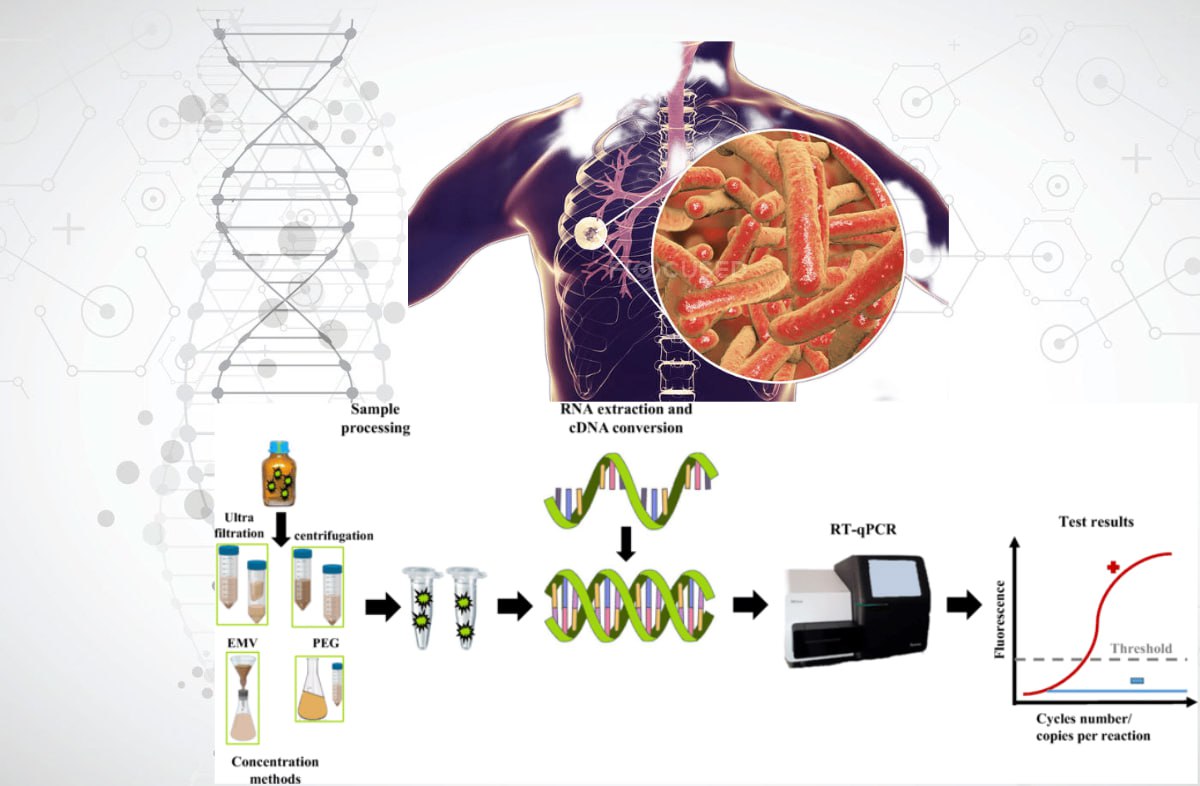
Results: it was determined that the modern methods for studying the genome of mycobacteria include amplification technologies (PCR analysis), hybridization, restriction, spoligotyping, sequencing, and their various combinations. The main methods are standard and modified protocols of PCR (RAPD-PCR, AP-PCR, rep-PCR, Real-time PCR, Inverse PCR, TB-LAMP, HIP, LM-PCR). Genomic Restriction Analysis can be used in studies of MTBC and NTM strains (RFLP, AFLP analysis, MIRU-VNTR genotyping). The most effective method for genome analysis is WGS. Complex methods that utilize a combination of molecular technologies allow for the direct detection of mycobacteria in clinical samples.
Conclusions: the widespread application of genomic technologies in the study of mycobacteria will contribute to the effective implementation of the global WHO strategy for the prevention, treatment, and control of tuberculosis and mycobacteriosis
References
- Global tuberculosis report 2020 (2020). World Health Organization, 208. Available at: https://apps.who.int/iris/handle/10665/336069
- Global tuberculosis report 2022 (2022). World Health Organization, 51. Available at: https://apps.who.int/iris/handle/10665/363752
- Cole, S. T., Brosch, R., Parkhill, J., Garnier, T., Churcher, C., Harris, D. et al. (1998). Deciphering the biology of Mycobacterium tuberculosis from the complete genome sequence. Nature, 393 (6685), 537–544. doi: https://doi.org/10.1038/31159
- Global tuberculosis report 2019 (2019). World Health Organization, 283. Available at: https://apps.who.int/iris/handle/10665/329368
- Barbova, A. I., Zhurilo, O. A., Trofimova, P. S., Mironchenko, S. V. (2019). Experience of selection, indication and identication of non-tuberculous mycobacteria, got during I National research on study of distribution of drugresistant of tuberculosis in Ukraine. Tuberculosis, Lung Diseases, HIV Infection, 2 (37), 63–71. doi: https://doi.org/10.30978/tb2019-2-63
- Yang, S., Rothman, R. E. (2004). PCR-based diagnostics for infectious diseases: uses, limitations, and future applications in acute-care settings. The Lancet Infectious Diseases, 4 (6), 337–348. doi: https://doi.org/10.1016/s1473-3099(04)01044-8
- Clark, D. P., Pazdernik, N. J. (2016). DNA Synthesis In Vivo and In Vitro. Biotechnology. Applying the Genetic Revolution, 97–130. doi: https://doi.org/10.1016/b978-0-12-385015-7.00004-1
- Nurwidya, F., Handayani, D., Burhan, E., Yunus, F. (2018). Molecular Diagnosis of Tuberculosis. Chonnam Medical Journal, 54 (1). doi: https://doi.org/10.4068/cmj.2018.54.1.1
- Chia, J.-H., Wu, T.-L., Su, L.-H., Kuo, A.-J., Lai, H.-C. (2012). Direct identification of mycobacteria from smear-positive sputum samples using an improved multiplex polymerase chain reaction assay. Diagnostic Microbiology and Infectious Disease, 72 (4), 340–349. doi: https://doi.org/10.1016/j.diagmicrobio.2011.12.008
- Van Crevel, R., Hill, P. C. (2017). Tuberculosis. SECTION 2 Syndromes by Body System: The Respiratory System. Infectious Diseases, Vol. 1, 271–284. doi: https://doi.org/10.1016/b978-0-7020-6285-8.00031-9
- Percival, S. L., Williams, D. W. (2014). Mycobacterium. Microbiology of Waterborne Diseases. Microbiological Aspects and Risks, 177–207. doi: https://doi.org/10.1016/B978-0-12-415846-7.00009-3
- Shapovalova, O. V., Pozmogova, S. A., Zavgorodniy, А. І. (2022). Molecular technologies of mycobacterial research. Annals of Mechnikov Institute, 1, 9–20. doi: https://doi.org/10.5281/zenodo.7721843
- Molecular assays intended as initial tests for the diagnosis of pulmonary and extrapulmonary TB and rifampicin resistance in adults and children: rapid communication (2020). World Health Organization, 8. Available at: https://apps.who.int/iris/handle/10665/330395
- Kohli, M., Schiller, I., Dendukuri, N., Yao, M., Dheda, K., Denkinger, C. M., Schumacher, S. G., Steingart, K. R. (2021). Xpert MTB/RIF Ultra and Xpert MTB/RIF assays for extrapulmonary tuberculosis and rifampicin resistance in adults. Cochrane Database of Systematic Reviews, 2021 (1). doi: https://doi.org/10.1002/14651858.cd012768.pub3
- Tuberculosis laboratory biosafety manual (2012). World Health Organization, 50. doi: https://apps.who.int/iris/handle/10665/77949
- Pro zatverdzhennia derzhavnykh sanitarnykh norm i pravyl "Orhanizatsiia roboty laboratorii pry doslidzhenni materialu, shcho mistyt biolohichni patohenni ahenty I-IV hrup patohennosti molekuliarno-henetychnymy metodamy" (2008). Hakaz MOZU No. 26. 24.01.2008. Available at: https://zakon.rada.gov.ua/laws/show/z0088-08#Text
- Gerilovich, A. P., Stegnіi, B. T., Zavgorodnіi, A. І., Vlіzlo, V. V. et al. (2014). Molekuliarno-genetichnі metodi dіagnostiki u veterinarnіi meditcinі ta bіotekhnologіi. Kyiv: ST-Druk, 286.
- Chauhan, T. (2019). Inverse PCR: Principle, Procedure, Protocol and Applications. PCR Technology. Genetic Education. Available at: https://geneticeducation.co.in/inverse-pcr-principle-procedure-protocol-and-applications/#google_vignette
- Notomi, T., Mori, Y., Tomita, N., Kanda, H. (2015). Loop-mediated isothermal amplification (LAMP): principle, features, and future prospects. Journal of Microbiology, 53 (1), 1–5. doi: https://doi.org/10.1007/s12275-015-4656-9
- Notomi, H., Okayama, H., Masubuchi, H., Yonekawa, T., Watanabe, K., Amino, N., Hase, T. (2000). Loop-mediated isothermal amplification of DNA. Nucleic Acids Research, 28 (12), 63e–663. doi: https://doi.org/10.1093/nar/28.12.e63
- Kim, J., Park, B. G., Lim, D. H., Jang, W. S., Nam, J., Mihn, D.-C., Lim, C. S. (2021). Development and evaluation of a multiplex loop-mediated isothermal amplification (LAMP) assay for differentiation of Mycobacterium tuberculosis and non-tuberculosis mycobacterium in clinical samples. PLOS ONE, 16 (1), e0244753. doi: https://doi.org/10.1371/journal.pone.0244753
- Molecular Test "LAMP". Available at: https://www.eiken.co.jp/en/products/lamp/
- Innovative Tool "TB LAMP". Available at: https://www.eiken.co.jp/en/ourfields/infection/tb_diagnosis/
- The use of loop-mediated isothermal amplification (TB-LAMP) for the diagnosis of pulmonary tuberculosis: policy guidance (2016). World Health Organization. Available at: https://apps.who.int/iris/handle/10665/249154
- Caulfield, A. J., Wengenack, N. L. (2016). Diagnosis of active tuberculosis disease: From microscopy to molecular techniques. Journal of Clinical Tuberculosis and Other Mycobacterial Diseases, 4, 33–43. doi: https://doi.org/10.1016/j.jctube.2016.05.005
- Line probe assays for detection of drug-resistant tuberculosis: interpretation and reporting manual for laboratory staff and clinicians (2022). World Health Organization, 31. https://apps.who.int/iris/handle/10665/354240
- Freeman, W. M., Robertson, D. J., Vrana, K. E. (2000). Fundamentals of DNA Hybridization Arrays for Gene Expression Analysis. BioTechniques, 29 (5), 1042–1055. doi: https://doi.org/10.2144/00295rv01
- Manual for selection of molecular WHO-recommended rapid diagnostic tests for detection of tuberculosis and drug-resistant tuberculosis (2022). World Health Organization, 29. https://apps.who.int/iris/handle/10665/353596
- Talbot, E. A., Raffa, B. J. (2015). Mycobacterium tuberculosis. Molecular Medical Microbiology, Vol. 3, 1637–1653. doi: https://doi.org/10.1016/B978-0-12-397169-2.00092-5
- Adam, M. A. M., Hamdan Ali, H. M., Khalil, E. A. G. (2019). Diagnostic predictive values of the hain genotype MTBDRsl assay in mycobacterial strains isolated from Sudan. Pan African Medical Journal, 32. doi: https://doi.org/10.11604/pamj.2019.32.124.12762
- Ou, X., Li, Q., Su, D., Xia, H., Wang, S., Zhao, B., Zhao, Y. (2020). A pilot study: VereMTB detection kit for rapid detection of multidrug-resistant mycobcterium tuberculosis in clinical sputum samples. PLOS ONE, 15 (3), e0228312. doi: https://doi.org/10.1371/journal.pone.0228312
- Algorithm for laboratory diagnosis and treatment-monitoring of pulmonary tuberculosis and drug-resistant tuberculosis using state-of-the-art rapid molecular diagnostic technologies: expert opinion of the European Tuberculosis Laboratory Initiative core group members for the WHO European Region (2017). World Health Organization. Regional Office for Europe. Available at: https://apps.who.int/iris/handle/10665/344108
- The use of molecular line probe assay for the detection of resistance to isoniazid and rifampicin: policy update (2016). World Health Organization, 56. https://apps.who.int/iris/handle/10665/250586
- Roychowdhury, T., Mandal, S., Bhattacharya, A. (2015). Analysis of IS6110 insertion sites provide a glimpse into genome evolution of Mycobacterium tuberculosis. Scientific Reports, 5 (1). doi: https://doi.org/10.1038/srep12567
- Jagielski, T., van Ingen, J., Rastogi, N., Dziadek, J., Mazur, P. K., Bielecki, J. (2014). Current Methods in the Molecular Typing of Mycobacterium tuberculosisand Other Mycobacteria. BioMed Research International, 2014, 1–21. doi: https://doi.org/10.1155/2014/645802
- Jagielski, T., Minias, A., van Ingen, J., Rastogi, N., Brzostek, A., Żaczek, A., Dziadek, J. (2016). Methodological and Clinical Aspects of the Molecular Epidemiology of Mycobacterium tuberculosis and Other Mycobacteria. Clinical Microbiology Reviews, 29 (2), 239–290. doi: https://doi.org/10.1128/cmr.00055-15
- Goulding, J. N., Stanley, J., Saunders, N., Arnold, C. (2000). Genome-Sequence-Based Fluorescent Amplified-Fragment Length Polymorphism Analysis of Mycobacterium tuberculosis. Journal of Clinical Microbiology, 38 (3), 1121–1126. doi: https://doi.org/10.1128/jcm.38.3.1121-1126.2000
- Kassama, Y., Shemko, M., Shetty, N., Fang, Z., MacIntire, G., Gant, V. et al. (2006). An Improved Fluorescent Amplified Fragment Length Polymorphism Method for Typing Mycobacterium tuberculosis. Journal of Clinical Microbiology, 44 (1), 288–289. doi: https://doi.org/10.1128/jcm.44.1.288-289.2006
- Varun, C. N. (2014). Clustered Regularly Interspaced Short Palindromic Repeats – Crispr. MICROBOIDS. Available at: https://varuncnmicro.blogspot.com/2014/02/clustered-regularly-interspaced-short.html
- Demay, C., Liens, B., Burguière, T., Hill, V., Couvin, D., Millet, J. et al. (2012). SITVITWEB – A publicly available international multimarker database for studying Mycobacterium tuberculosis genetic diversity and molecular epidemiology. Infection, Genetics and Evolution, 12 (4), 755–766. doi: https://doi.org/10.1016/j.meegid.2012.02.004
- Brudey, K., Driscoll, J. R., Rigouts, L., Prodinger, W. M., Gori, A., Al-Hajoj, S. A. et al. (2006). Mycobacterium tuberculosis complex genetic diversity: mining the fourth international spoligotyping database (SpolDB4) for classification, population genetics and epidemiology. BMC Microbiology, 6 (1). doi: https://doi.org/10.1186/1471-2180-6-23
- Bouakaze, C., Keyser, C., Gonzalez, A., Sougakoff, W., Veziris, N., Dabernat, H. et al. (2011). Matrix-Assisted Laser Desorption Ionization-Time of Flight Mass Spectrometry-Based Single Nucleotide Polymorphism Genotyping Assay Using iPLEX Gold Technology for Identification of Mycobacterium tuberculosis Complex Species and Lineages. Journal of Clinical Microbiology, 49 (9), 3292–3299. doi: https://doi.org/10.1128/jcm.00744-11
- Burian, A. N., Zhao, W., Lo, T., Thurtle‐Schmidt, D. M. (2021). Genome sequencing guide: An introductory toolbox to whole‐genome analysis methods. Biochemistry and Molecular Biology Education, 49 (5), 815–825. doi: https://doi.org/10.1002/bmb.21561
- Bacterial Diversity Metadatabase BacDive. Available at: https://bacdive.dsmz.de/about
- The use of next-generation sequencing technologies for the detection of mutations associated with drug resistance in Mycobacterium tuberculosis complex: technical guide (2018). World Health Organization, 112. Available at: https://apps.who.int/iris/handle/10665/274443
- Zhou, X., Wu, H., Ruan, Q., Jiang, N., Chen, X., Shen, Y. et al. (2019). Clinical Evaluation of Diagnosis Efficacy of Active Mycobacterium tuberculosis Complex Infection via Metagenomic Next-Generation Sequencing of Direct Clinical Samples. Frontiers in Cellular and Infection Microbiology, 9. doi: https://doi.org/10.3389/fcimb.2019.00351
- Anochie, P. I., Onyeneke, E. C., Ogu, A. C., Onyeozirila, A. C., Aluru, S., Onyejepu, N. et al. (2012). Recent advances in the diagnosis of Mycobacterium tuberculosis. GERMS, 2 (3), 110–120. doi: https://doi.org/10.11599/germs.2012.1021
- Leão, S. C., Martin, A., Meija M, G. I., Palomino, J. C., Robledo, J., da Silva Telles, M. A., Portaels, F. (2004). Practical handbook for the phenotypic and genotypic identification of mycobacteria. Available at: http://hdl.handle.net/1854/LU-7188307
- Schlossberg, D. (Ed.) (2017). Tuberculosis and nontuberculous mycobacterial infections. Washington: ASM Press, 800. doi: https://doi.org/10.1128/9781555819866
- Schaaf, H.S., Zumla, A. (Eds.) (2009). Tuberculosis: A Comprehensive Clinical Reference. Philadelphia: Saunders Elsevier, 1046.
- Balasingham, S. V., Davidsen, T., Szpinda, I., Frye, S. A., Tønjum, T. (2009). Molecular diagnostics in tuberculosis: basis and implications for therapy. Molecular Diagnosis & Therapy, 13 (3), 137–151. doi: https://doi.org/10.1007/bf03256322
- Scarparo, C., Piccoli, P., Rigon, A., Ruggiero, G., Scagnelli, M., Piersimoni, C. (2000). Comparison of enhanced Mycobacterium tuberculosis amplified direct test with COBAS AMPLICOR Mycobacterium tuberculosis assay for direct detection of Mycobacterium tuberculosis complex in respiratory and extrapulmonary specimens. Journal of Clinical Microbiology, 38 (4), 1559–1562. doi: https://doi.org/10.1128/jcm.38.4.1559-1562.2000
- da Silva, D.A., de Pina, L.C., Rêgo, A.M., Ferreira, N.V., Redner, P., Caetano, L., Antunes, M.; Tang, Y.-W., Stratton, C. W. (Eds.) (2018). Advances in the Diagnosis of Mycobacterium tuberculosis Infection. Advanced Techniques in Diagnostic Microbiology. Vol. 2: Applications. Springer Nature Switzerland AG. 101–136. doi: https://doi.org/10.1007/978-3-319-95111-9_4
- European Centre for Disease Prevention and Control. Handbook on tuberculosis laboratory diagnostic methods in the European Union – Updated 2018 (2018). Stockholm: ECDC. Available at: https://www.ecdc.europa.eu/en/publications-data/handbook-tuberculosis-laboratory-diagnostic-methods-european-union-updated-2018
Downloads
Published
How to Cite
Issue
Section
License
Copyright (c) 2023 Olga Shapovalova, Olena Koshova, Nataliia Filimonova

This work is licensed under a Creative Commons Attribution 4.0 International License.
Our journal abides by the Creative Commons CC BY copyright rights and permissions for open access journals.
Authors, who are published in this journal, agree to the following conditions:
1. The authors reserve the right to authorship of the work and pass the first publication right of this work to the journal under the terms of a Creative Commons CC BY, which allows others to freely distribute the published research with the obligatory reference to the authors of the original work and the first publication of the work in this journal.
2. The authors have the right to conclude separate supplement agreements that relate to non-exclusive work distribution in the form in which it has been published by the journal (for example, to upload the work to the online storage of the journal or publish it as part of a monograph), provided that the reference to the first publication of the work in this journal is included.








