Бактеріальні плазміди: роль у розвитку стійкості до антимікробних сполук
DOI:
https://doi.org/10.15587/2519-8025.2024.311822Ключові слова:
мікробіологія, бактерії, плазміди, генетична мінливість та модіфікація, антибіотики, адаптація, резистентностьАнотація
Швидке зростання поширеності множинної лікарської стійкості патогенних мікроорганімів становить критичну загрозу громадському здоров’ю в усьому світі, що значно сприяє підвищенню смертності і захворюваності пацієнтів. Класичні засоби, які в минулому використовувалися для лікування, втрачають свою ефективність, більш того, багато з новіших доступних препаратів вже стали мішенями для бактеріальних механізмів резистентності. В результаті - лікування інфекцій ускладнюється, загальні витрати на лікування збільшуються.
Мета. В даній роботі ми поставили за мету оцінити роль плазмід у розвитку антибіотикорезистентності та обговорюємо різні механізми резистентності бактерій до антибіотиків, таких як ферментативна інактивація антибіотика, зменьшення проникності зовнішньої клітинної мембрани для антибіотика, модифікація мішені переважно за рахунок мутації, активний eфлюкс препарату з бактеріальної клітини за допомогою ферментативних помп.
Матеріали і методи: пошук джерел інформації здійснювався в базах даних PubMed, Medline, Web of Science, Google Scholar, а також електронних репозиторіях закладів вищої освіти та наукових установ України. Відібрано матеріали, пов’язані з технологією дослідження генетичної мінливості і модифікації бактерій та механізмів стійкості мікроорганізмів до антибіотиків.
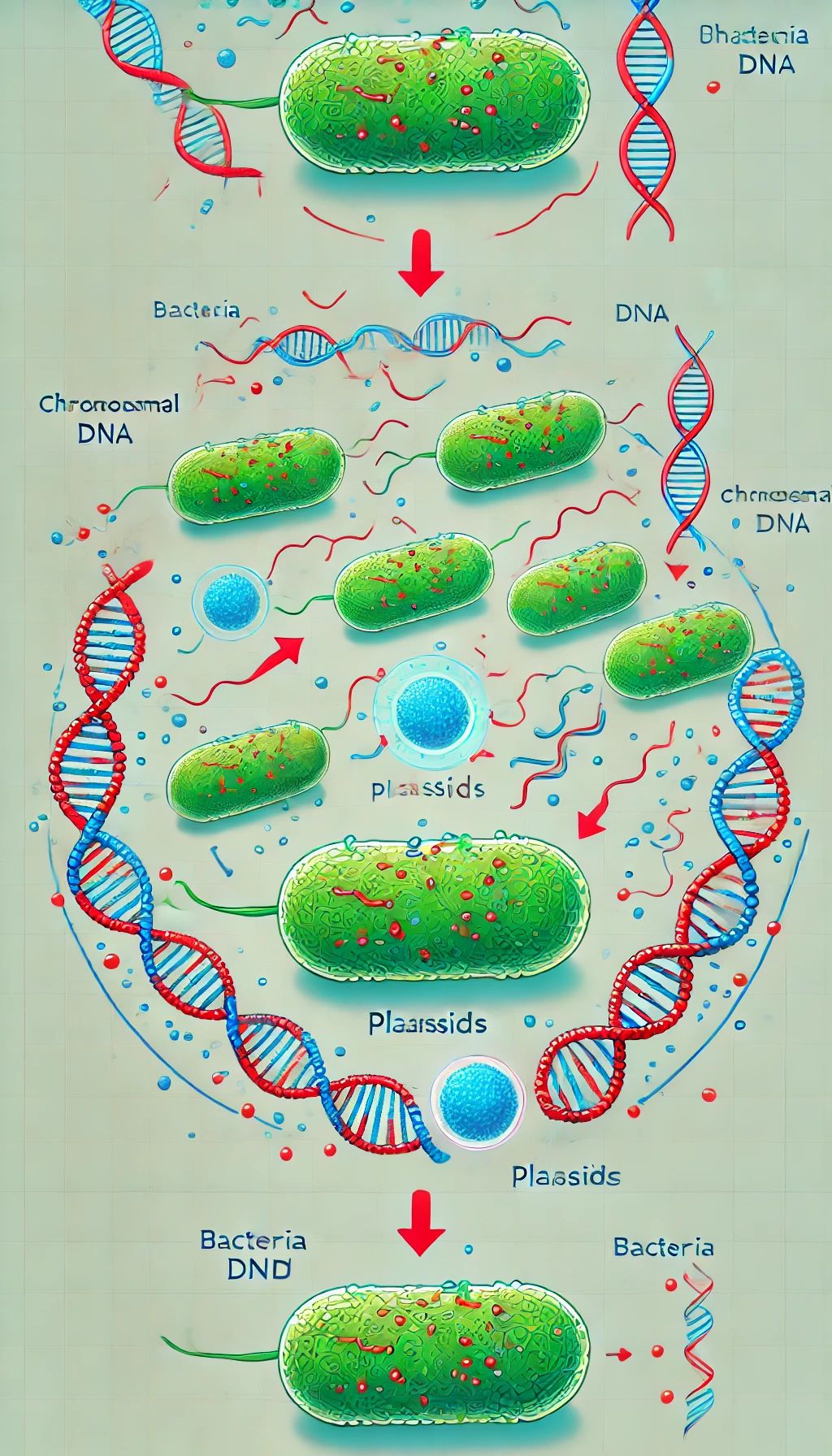
Результати: Внаслідок еволюційного розвитку бактерії здобули дві окремі генетичні системи - хромосомна ДНК і позахромосомні, самовідтворювані генетичні елементи, які називають плазмідами.
Саме бактеріальні плазміди відіграють ключову роль у дифузії генів специфічної стійкості, зокрема до антибіотиків. Плазміди є необов’язковими частинами бактерій і представлені дволанцюговими, кільцевими або лінійними молекулами ДНК, що здатні до автономної реплікації, завдяки чому бактерії пристосовуються у навколишньому ворожому середовищі. На сьогодні найбільшу зацікавленість вчених викликають дві властивості бактерій, обумовлені плазмідами - антибіотикорезистентність та біоремедіація. Остання обумовлює виживання та розвиток бактерій в різних несприятливих умовах, включаючи розвиток стійкості до забруднювачів, здатність розкладати різні хімічні сполуки або адаптацію до нових екологічних ніш.
Висновки. Плазміди несуть гени деградації ксенобіотиків і стійкості до важких металів, що робить їх корисними для біоремедіації токсичних хімічних речовин екологічно безпечним способом. Проте, таки властивості, як стійкість до антибіотиків, є наслідком надмірного та безконтрольного застосування цих ліків у медицині, ветеринарії, сільському господарстві тощо. У таких умовах відбувається природний відбір тих штамів патогенних бактерій, які є носіями R-плазмід
Посилання
- Banu, H., Prasad, K. P. (2017). Role of Plasmids in Microbiology. Journal of Aquaculture Research & Development, 8 (1). https://doi.org/10.4172/2155-9546.1000466
- Actis, L. A., Tolmasky, M. E., Crosa, J. H. (1999). Bacterial plasmids replication of extrachromosomal genetic elements encoding resistance to antimicrobial compounds. Frontiers in Bioscience, 3 (4), d43–62. https://doi.org/10.2741/a410
- Leplae, R., Hebrant, A., Wodak, S. J., Toussaint, A. (2004). ACLAME: A CLAssification of Mobile genetic Elements. Nucleic Acids Research, 32 (1), D45–D49. https://doi.org/10.1093/nar/gkh084
- Carattoli, A. (2011). Plasmids in Gram negatives: Molecular typing of resistance plasmids. International Journal of Medical Microbiology, 301 (8), 654–658. https://doi.org/10.1016/j.ijmm.2011.09.003
- Mc Ginty, S. (2012).The role of horizontal gene transfer in microbial social evolution [Doctoral dissertation, University of Zurich].
- Barlow, M. (2009). What Antimicrobial Resistance Has Taught Us About Horizontal Gene Transfer. Horizontal Gene Transfer, 397–411. https://doi.org/10.1007/978-1-60327-853-9_23
- Helinski, D. R. (2022). A Brief History of Plasmids. EcoSal Plus, 10 (1). https://doi.org/10.1128/ecosalplus.esp-0028-2021
- Hayes, W. (1968). The genetics of bacteria and their viruses. New York: John Wiley & Sons Inc.
- LEDERBERG, J., TATUM, E. L. (1946). Gene Recombination in Escherichia Coli. Nature, 158 (4016), 558–562. https://doi.org/10.1038/158558a0
- Watson, J. D., Crick, F. H. C. (1953). Molecular Structure of Nucleic Acids: A Structure for Deoxyribose Nucleic Acid. Nature, 171 (4356), 737–738. https://doi.org/10.1038/171737a0
- Kornberg, A. (1960). Biologic Synthesis of Deoxyribonucleic Acid. Science, 131 (3412), 1503–1508. https://doi.org/10.1126/science.131.3412.1503
- Watanabe, T. (1963). Infective heredity of multiple drug resistance in bacteria. Bacteriological Reviews, 27 (1), 87–115. https://doi.org/10.1128/br.27.1.87-115.1963
- Smillie, C., Garcillán-Barcia, M. P., Francia, M. V., Rocha, E. P. C., de la Cruz, F. (2010). Mobility of Plasmids. Microbiology and Molecular Biology Reviews, 74 (3), 434–452. https://doi.org/10.1128/mmbr.00020-10
- Wang, Z., Jin, L., Yuan, Z., Węgrzyn, G., Węgrzyn, A. (2009). Classification of plasmid vectors using replication origin, selection marker and promoter as criteria. Plasmid, 61 (1), 47–51. https://doi.org/10.1016/j.plasmid.2008.09.003
- Anthony, K. G., Sherburne, C., Sherburne, R., Frost, L. S. (1994). The role of the pilus in recipient cell recognition during bacterial conjugation mediated by F‐like plasmids. Molecular Microbiology, 13 (6), 939–953. https://doi.org/10.1111/j.1365-2958.1994.tb00486.x
- Zverev, V. V., Kuzmin, N. P., Zuyeva, L. A., Burova, E. I., Alexandrov, A. A., Khmel, I. A. (1984). Regions of homology in small colicinogenic plasmids. Plasmid, 12 (3), 203–205. https://doi.org/10.1016/0147-619x(84)90045-3
- Mahajan, P., Kumar, M., Bhalla, G. S., Tandel, K. (2024). Plasmid-based replicon typing: Useful tool in demonstrating the silent pandemic of plasmid-mediated multi-drug resistance in Enterobacterales. Medical Journal Armed Forces India. https://doi.org/10.1016/j.mjafi.2024.02.004
- Chun, D., Cho, D. T., Seol, S. Y., Suh, M. H., Lee, Y. C. (1984). R plasmids conferring multiple drug resistance from shigella isolated in Korea. Journal of Hygiene, 92 (2), 153–160. https://doi.org/10.1017/s0022172400064160
- Johnson, J., Warren, R. L., Branstrom, A. A. (1991). Effects of FP2 and a mercury resistance plasmid from Pseudomonas aeruginosa PA103 on exoenzyme production. Journal of Clinical Microbiology, 29 (5), 940–944. https://doi.org/10.1128/jcm.29.5.940-944.1991
- Garcillán-Barcia, M. P., Alvarado, A., de la Cruz, F. (2011). Identification of bacterial plasmids based on mobility and plasmid population biology. FEMS Microbiology Reviews, 35 (5), 936–956. https://doi.org/10.1111/j.1574-6976.2011.00291.x
- Romaniuk, L. B., Kravets, N. Y., Klymniuk, S. I., Kopcha, V. S., Dronova, O. Y. (2020). Antybiotykorezystentnist umovno-patohennykh mikroorhanizmiv: aktualnist, umovy vynyknennia, shliakhy podolannia. Infektsiini khvoroby, 4, 63–71. https://doi.org/10.11603/1681-2727.2019.4.10965
##submission.downloads##
Опубліковано
Як цитувати
Номер
Розділ
Ліцензія
Авторське право (c) 2024 Olena Koshova, Nataliia Filimonova, Larysa Mozghova, Iryna Tishchenko

Ця робота ліцензується відповідно до Creative Commons Attribution 4.0 International License.
Наше видання використовує положення про авторські права Creative Commons CC BY для журналів відкритого доступу.
Автори, які публікуються у цьому журналі, погоджуються з наступними умовами:
1. Автори залишають за собою право на авторство своєї роботи та передають журналу право першої публікації цієї роботи на умовах ліцензії Creative Commons CC BY, котра дозволяє іншим особам вільно розповсюджувати опубліковану роботу з обов'язковим посиланням на авторів оригінальної роботи та першу публікацію роботи у цьому журналі.
2. Автори мають право укладати самостійні додаткові угоди щодо неексклюзивного розповсюдження роботи у тому вигляді, в якому вона була опублікована цим журналом (наприклад, розміщувати роботу в електронному сховищі установи або публікувати у складі монографії), за умови збереження посилання на першу публікацію роботи у цьому журналі.








