Targeted structural design of molecular scaffolds for dual-action peptidomimetic inhibitors against SARS-CoV-2 MPRO and PLPRO
DOI:
https://doi.org/10.15587/2519-4852.2025.337951Keywords:
SARS-CoV-2, Mpro protease, PLpro protease, peptidomimetics, dual inhibitors, molecular dockingAbstract
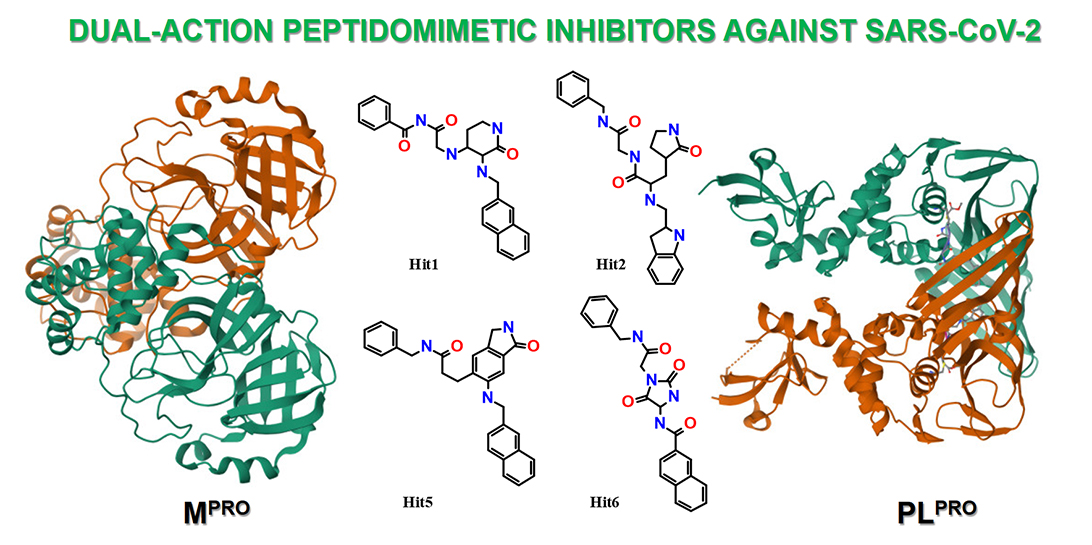
The two proteases of SARS-CoV-2 coronavirus – the main protease (Mpro or 3CLpro) and the papain-like protease (PLpro) – are essential enzymes required for the successful replication of the virus within cells. Both proteases have become major targets in the development of antiviral drugs against SARS-CoV-2. The potential to achieve a dual inhibitory effect has sparked significant interest in creating dual inhibitors as complex therapeutic agents for this virus. In this article, we discuss the development and in silico evaluation of a series of new peptidomimetic molecules designed as dual-action inhibitors of both SARS-CoV-2 Mpro and PLpro, along with their synthesis. We implemented a combined approach that began with developing a basic molecular model, considering the substrate specificity of the active centers of each protease. Through rational in silico design, we created a series of peptidomimetics. Further analysis of how these compounds bound to the active sites of both proteases enabled us to identify several new structural hits, including hydantoin derivatives, as potential dual inhibitors of Mpro and PLpro.
The aim of the study. This study aims to establish a common molecular framework for designing dual-action inhibitors targeting the SARS-CoV-2 Mpro and PLpro proteases. The research includes receptor-oriented molecular docking, in silico optimization, and the selection and synthesis of the most active candidate structures for further in vitro experimental studies.
Materials and methods. LigandScout 4.5 software is used for 3D pharmacophore analysis, visualization, and molecular docking. AutoDock Vina 1.1 provides tools for molecular docking. The PLIP (Protein-Ligand Interaction Profiler) web servers are utilized to study molecular binding mechanisms. DataWarrior 6.0 software helps create a library of molecular structures, calculate physicochemical properties, and analyze molecular frameworks. SwissADME web server is used to predict ADME parameters and assess the pharmacokinetic properties of small molecules as potential drugs.
Results. We analyzed the substrate specificity of the binding sites of the Mpro and PLpro proteases, which enabled us to identify a common amino acid sequence containing shared recognition elements for both proteases. By rationally modifying the functional groups in this initial base structure, utilizing the principle of isosteric replacement and incorporating non-classical bioisosteres, we developed a series of peptidomimetic frameworks. Molecular docking conducted at the active sites of both Mpro and PLpro, along with the assessment of their binding energy values (in kcal/mol), identified several structures with potential for dual inhibition. Notably, hydantoin derivatives demonstrated the strongest binding affinity to the active sites of both proteases.
Conclusions. We have identified promising peptidomimetic molecular structures that demonstrate dual inhibitory activity against the SARS-CoV-2 proteases through in silico analysis. Specifically, we discovered a novel class of hydantoin derivatives that act as inhibitors for both SARS-CoV-2 Mpro and PLpro. The synthesis methods we developed allow for the preparation of these compounds for further in vitro studies
Supporting Agency
- Grant No. 96/0062 (2021.01/0062) “Molecular design, synthesis and screening of new potential antiviral pharmaceutical ingredients for the treatment of infectious diseases COVID-19” from the National Research Foundation of Ukraine
References
- Yevsieieva, L. V., Lohachova, K. O., Kyrychenko, A., Kovalenko, S. M., Ivanov, V. V., Kalugin, O. N. (2023). Main and papain-like proteases as prospective targets for pharmacological treatment of coronavirus SARS-CoV-2. RSC Advances, 13 (50), 35500–35524. https://doi.org/10.1039/d3ra06479d
- Capasso, C., Nocentini, A., Supuran, C. T. (2020). Protease inhibitors targeting the main protease and papain-like protease of coronaviruses. Expert Opinion on Therapeutic Patents, 31 (4), 309–324. https://doi.org/10.1080/13543776.2021.1857726
- Wang, Y., Xu, B., Ma, S., Wang, H., Shang, L., Zhu, C., Ye, S. (2022). Discovery of SARS-CoV-2 3CLPro Peptidomimetic Inhibitors through the Catalytic Dyad Histidine-Specific Protein-Ligand Interactions. International Journal of Molecular Sciences, 23 (4), 2392. https://doi.org/10.3390/ijms23042392
- Kerti, L., Frecer, V. (2024). Design of inhibitors of SARS-CoV-2 papain-like protease deriving from GRL0617: Structure–activity relationships. Bioorganic & Medicinal Chemistry, 113, 117909. https://doi.org/10.1016/j.bmc.2024.117909
- Lohachova, K. O., Kyrychenko, A., Kalugin, O. N. (2025). Benchmarking biomolecular force fields for molecular dynamics simulations of native fold and enzymatic activity of SARS-CoV-2 papain-like protease. Heliyon, 11 (12), e43578. https://doi.org/10.1016/j.heliyon.2025.e43578
- Garnsey, M. R., Robinson, M. C., Nguyen, L. T., Cardin, R., Tillotson, J., Mashalidis, E. et al. (2024). Discovery of SARS-CoV-2 papain-like protease (PLpro) inhibitors with efficacy in a murine infection model. bioRxiv. https://doi.org/10.1101/2024.01.26.577395
- Yevsieieva, L., Trostianko, P., Kyrychenko, A., Ivanov, V., Kovalenko, S., Kalugin, O. (2024). Design of non-covalent dual-acting inhibitors for proteases MPRO and PLPRO of coronavirus SARS-CoV-2 through evolutionary library generation, pharmacophore profile matching, and molecular docking calculations. ScienceRise: Pharmaceutical Science, 6 (52), 15–26. https://doi.org/10.15587/2519-4852.2024.313808
- Ma, C., Sacco, M. D., Xia, Z., Lambrinidis, G., Townsend, J. A., Hu, Y. et al. (2021). Discovery of SARS-CoV-2 Papain-like Protease Inhibitors through a Combination of High-Throughput Screening and a FlipGFP-Based Reporter Assay. ACS Central Science, 7 (7), 1245–1260. https://doi.org/10.1021/acscentsci.1c00519
- Lucas, S. C. C., Blackwell, J. H., Hewitt, S. H., Semple, H., Whitehurst, B. C., Xu, H. (2024). Covalent hits and where to find them. SLAS Discovery, 29 (3), 100142. https://doi.org/10.1016/j.slasd.2024.01.003
- Ivanov, V., Lohachova, K., Kolesnik, Y., Zakharov, A., Yevsieieva, L., Kyrychenko, A. et al. (2023). Recent advances in computational drug discovery for therapy against coronavirus SARS-CoV-2. ScienceRise: Pharmaceutical Science, 6 (46), 4–24. https://doi.org/10.15587/2519-4852.2023.290318
- Zhang, L., Lin, D., Sun, X., Curth, U., Drosten, C., Sauerhering, L. et al. (2020). Crystal structure of SARS-CoV-2 main protease provides a basis for design of improved α-ketoamide inhibitors. Science, 368 (6489), 409–412. https://doi.org/10.1126/science.abb3405
- Xia, Z., Sacco, M., Hu, Y., Ma, C., Meng, X., Zhang, F. et al. (2021). Rational Design of Hybrid SARS-CoV-2 Main Protease Inhibitors Guided by the Superimposed Cocrystal Structures with the Peptidomimetic Inhibitors GC-376, Telaprevir, and Boceprevir. ACS Pharmacology & Translational Science, 4 (4), 1408–1421. https://doi.org/10.1021/acsptsci.1c00099
- Citarella, A., Scala, A., Piperno, A., Micale, N. (2021). SARS-CoV-2 Mpro: A Potential Target for Peptidomimetics and Small-Molecule Inhibitors. Biomolecules, 11 (4), 607. https://doi.org/10.3390/biom11040607
- Bege, M., Borbás, A. (2024). The Design, Synthesis and Mechanism of Action of Paxlovid, a Protease Inhibitor Drug Combination for the Treatment of COVID-19. Pharmaceutics, 16 (2), 217. https://doi.org/10.3390/pharmaceutics16020217
- Lohachova, K. O., Sviatenko, A. S., Kyrychenko, A., Ivanov, V. V., Langer, T., Kovalenko, S. M., Kalugin, O. N. (2024). Computer-aided drug design of novel nirmatrelvir analogs inhibiting main protease of Coronavirus SARS-CoV-2. Journal of Applied Pharmaceutical Science, 14 (5), 232–239. https://doi.org/10.7324/japs.2024.158114
- Arafet, K., Serrano-Aparicio, N., Lodola, A., Mulholland, A. J., González, F. V., Świderek, K., Moliner, V. (2021). Mechanism of inhibition of SARS-CoV-2 Mpro by N3 peptidyl Michael acceptor explained by QM/MM simulations and design of new derivatives with tunable chemical reactivity. Chemical Science, 12 (4), 1433–1444. https://doi.org/10.1039/d0sc06195f
- Bauer, R. A. (2015). Covalent inhibitors in drug discovery: from accidental discoveries to avoided liabilities and designed therapies. Drug Discovery Today, 20 (9), 1061–1073. https://doi.org/10.1016/j.drudis.2015.05.005
- Singh, J., Petter, R. C., Baillie, T. A., Whitty, A. (2011). The resurgence of covalent drugs. Nature Reviews Drug Discovery, 10 (4), 307–317. https://doi.org/10.1038/nrd3410
- Schaefer, D., Cheng, X. (2023). Recent Advances in Covalent Drug Discovery. Pharmaceuticals, 16 (5), 663. https://doi.org/10.3390/ph16050663
- Hu, Y., Ma, C., Szeto, T., Hurst, B., Tarbet, B., Wang, J. (2021). Boceprevir, Calpain Inhibitors II and XII, and GC-376 Have Broad-Spectrum Antiviral Activity against Coronaviruses. ACS Infectious Diseases, 7 (3), 586–597. https://doi.org/10.1021/acsinfecdis.0c00761
- Kneller, D. W., Li, H., Phillips, G., Weiss, K. L., Zhang, Q., Arnould, M. A. et al. (2022). Covalent narlaprevir- and boceprevir-derived hybrid inhibitors of SARS-CoV-2 main protease. Nature Communications, 13 (1), 2268. https://doi.org/10.1038/s41467-022-29915-z
- Joyce, R. P., Hu, V. W., Wang, J. (2022). The history, mechanism, and perspectives of nirmatrelvir (PF-07321332): an orally bioavailable main protease inhibitor used in combination with ritonavir to reduce COVID-19-related hospitalizations. Medicinal Chemistry Research, 31 (10), 1637–1646. https://doi.org/10.1007/s00044-022-02951-6
- Tan, H., Hu, Y., Jadhav, P., Tan, B., Wang, J. (2022). Progress and Challenges in Targeting the SARS-CoV-2 Papain-like Protease. Journal of Medicinal Chemistry, 65 (11), 7561–7580. https://doi.org/10.1021/acs.jmedchem.2c00303
- Osipiuk, J., Azizi, S.-A., Dvorkin, S., Endres, M., Jedrzejczak, R., Jones, K. A. et al. (2021). Structure of papain-like protease from SARS-CoV-2 and its complexes with non-covalent inhibitors. Nature Communications, 12 (1), 743. https://doi.org/10.1038/s41467-021-21060-3
- Ton, A.-T., Pandey, M., Smith, J. R., Ban, F., Fernandez, M., Cherkasov, A. (2022). Targeting SARS-CoV-2 papain-like protease in the postvaccine era. Trends in Pharmacological Sciences, 43 (11), 906–919. https://doi.org/10.1016/j.tips.2022.08.008
- Rut, W., Lv, Z., Zmudzinski, M., Patchett, S., Nayak, D., Snipas, S. J. et al. (2020). Activity profiling and crystal structures of inhibitor-bound SARS-CoV-2 papain-like protease: A framework for anti–COVID-19 drug design. Science Advances, 6 (42), eabd4596. https://doi.org/10.1126/sciadv.abd4596
- Gao, X., Qin, B., Chen, P., Zhu, K., Hou, P., Wojdyla, J. A., Wang, M., Cui, S. (2021). Crystal structure of SARS-CoV-2 papain-like protease. Acta Pharmaceutica Sinica B, 11 (1), 237–245. https://doi.org/10.1016/j.apsb.2020.08.014
- Ullrich, S., Nitsche, C. (2022). SARS‐CoV‐2 Papain‐Like Protease: Structure, Function and Inhibition. ChemBioChem, 23 (19). https://doi.org/10.1002/cbic.202200327
- Yuan, S., Gao, X., Tang, K., Cai, J.-P., Hu, M., Luo, P. et al. (2022). Targeting papain-like protease for broad-spectrum coronavirus inhibition. Protein & Cell, 13 (12), 940–953. https://doi.org/10.1007/s13238-022-00909-3
- Wang, Q., Chen, G., He, J., Li, J., Xiong, M., Su, H. et al. (2023). Structure-Based Design of Potent Peptidomimetic Inhibitors Covalently Targeting SARS-CoV-2 Papain-like Protease. International Journal of Molecular Sciences, 24 (10), 8633. https://doi.org/10.3390/ijms24108633
- Yu, W., Zhao, Y., Ye, H., Wu, N., Liao, Y., Chen, N. et al. (2022). Structure-Based Design of a Dual-Targeted Covalent Inhibitor Against Papain-like and Main Proteases of SARS-CoV-2. Journal of Medicinal Chemistry, 65 (24), 16252–16267. https://doi.org/10.1021/acs.jmedchem.2c00954
- Santos, L. H., Kronenberger, T., Almeida, R. G., Silva, E. B., Rocha, R. E. O., Oliveira, J. C. et al. (2022). Structure-Based Identification of Naphthoquinones and Derivatives as Novel Inhibitors of Main Protease Mpro and Papain-like Protease PLpro of SARS-CoV-2. Journal of Chemical Information and Modeling, 62 (24), 6553–6573. https://doi.org/10.1021/acs.jcim.2c00693
- Reddy, A. S., Zhang, S. (2013). Polypharmacology: drug discovery for the future. Expert Review of Clinical Pharmacology, 6 (1), 41–47. https://doi.org/10.1586/ecp.12.74
- Sander, T., Freyss, J., von Korff, M., Rufener, C. (2015). DataWarrior: An Open-Source Program For Chemistry Aware Data Visualization And Analysis. Journal of Chemical Information and Modeling, 55 (2), 460–473. https://doi.org/10.1021/ci500588j
- Wolber, G., Langer, T. (2005). LigandScout: 3-D Pharmacophores Derived from Protein-Bound Ligands and Their Use as Virtual Screening Filters. Journal of Chemical Information and Modeling, 45 (1), 160–169. https://doi.org/10.1021/ci049885e
- Goodsell, D. S., Morris, G. M., Olson, A. J. (1996). Automated docking of flexible ligands: Applications of AutoDock. Journal of Molecular Recognition, 9(1), 1–5. https://doi.org/10.1002/(sici)1099-1352(199601)9:1<1::aid-jmr241>3.0.co;2-6
- Goodsell, D. S., Sanner, M. F., Olson, A. J., Forli, S. (2020). The AutoDock suite at 30. Protein Science, 30 (1), 31–43. https://doi.org/10.1002/pro.3934
- Kumar, T. D. A. (2022). Drug Design. A Conceptual Overview. Abingdon: CRC Press. https://doi.org/10.1201/9781003298755
- La Monica, G., Bono, A., Lauria, A., Martorana, A. (2022). Targeting SARS-CoV-2 Main Protease for Treatment of COVID-19: Covalent Inhibitors Structure-Activity Relationship Insights and Evolution Perspectives. Journal of Medicinal Chemistry, 65 (19), 12500–12534. https://doi.org/10.1021/acs.jmedchem.2c01005
- Bucherer, H. Th., Lieb, V. A. (1934). Über die Bildung substituierter Hydantoine aus Aldehyden und Ketonen. Synthese von Hydantoinen. Journal Für Praktische Chemie, 141 (1-2), 5–43. https://doi.org/10.1002/prac.19341410102
- Konnert, L., Lamaty, F., Martinez, J., Colacino, E. (2017). Recent Advances in the Synthesis of Hydantoins: The State of the Art of a Valuable Scaffold. Chemical Reviews, 117 (23), 13757–13809. https://doi.org/10.1021/acs.chemrev.7b00067
- Kalník, M., Gabko, P., Bella, M., Koóš, M. (2021). The Bucherer-Bergs Multicomponent Synthesis of Hydantoins – Excellence in Simplicity. Molecules, 26 (13), 4024. https://doi.org/10.3390/molecules26134024
- Park, E. J., Lee, S., Chang, S. (2010). Acetone Cyanohydrin as a Source of HCN in the Cu-Catalyzed Hydrocyanation of α-Aryl Diazoacetates. The Journal of Organic Chemistry, 75 (8), 2760–2762. https://doi.org/10.1021/jo100356d
- Cho, S., Kim, S.-H., Shin, D. (2019). Recent applications of hydantoin and thiohydantoin in medicinal chemistry. European Journal of Medicinal Chemistry, 164, 517–545. https://doi.org/10.1016/j.ejmech.2018.12.066
- Ölmez, N. A., Waseer, F. (2020). New Potential Biologically Active Compounds: Synthesis and Characterization of Urea and Thiourea Derivativpes Bearing 1,2,4-oxadiazole Ring. Current Organic Synthesis, 17 (7), 525–534. https://doi.org/10.2174/1570179417666200417112106
- Zhu, L., Zeng, H., Liu, D., Fu, Y., Wu, Q., Song, B., Gan, X. (2020). Design, synthesis, and biological activity of novel 1,2,4-oxadiazole derivatives. BMC Chemistry, 14 (1), 68. https://doi.org/10.1186/s13065-020-00722-1
- Perekhoda, L., Suleiman, M., Podolsky, I., Semenets, A., Kobzar, N., Yaremenko, V. et al. (2024). Synthesis and nootropic activity prediction of some 4-(aminomethyl)-1-benzylpyrrolidin-2-one derivatives structurally related with nebracetam. ScienceRise: Pharmaceutical Science, 4 (50), 23–34. https://doi.org/10.15587/2519-4852.2024.310731
- Semenets, A. P., Suleiman, M. M., Fedosov, A. I., Shtrygol, S. Y., Havrylov, I. O., Mishchenko, M. V. et al. (2022). Synthesis, docking, and biological evaluation of novel 1-benzyl-4-(4-(R)-5-sulfonylidene-4,5-dihydro-1H-1,2,4-triazol-3-yl)pyrrolidin-2-ones as potential nootropic agents. European Journal of Medicinal Chemistry, 244, 114823. https://doi.org/10.1016/j.ejmech.2022.114823
- Shintani, Y., Kato, K., Kawami, M., Takano, M., Kumamoto, T. (2021). Direct N1-Selective Alkylation of Hydantoins Using Potassium Bases. Chemical and Pharmaceutical Bulletin, 69 (4), 407–410. https://doi.org/10.1248/cpb.c20-00857
Downloads
Published
How to Cite
Issue
Section
License
Copyright (c) 2025 Larysa Yevsieieva, Alexander Kyrychenko, Pavlo Trostianko, Volodymyr Ivanov, Sergiy M. Kovalenko, Oleg Kalugin

This work is licensed under a Creative Commons Attribution 4.0 International License.
Our journal abides by the Creative Commons CC BY copyright rights and permissions for open access journals.






