Автоматизоване видобування ключових параметрів та виявлення невідповідностей у клінічній документації за допомогою великих мовних моделей
DOI:
https://doi.org/10.15587/1729-4061.2025.337915Ключові слова:
клінічні випробування, великі мовні моделі, клінічна документація, видобування данихАнотація
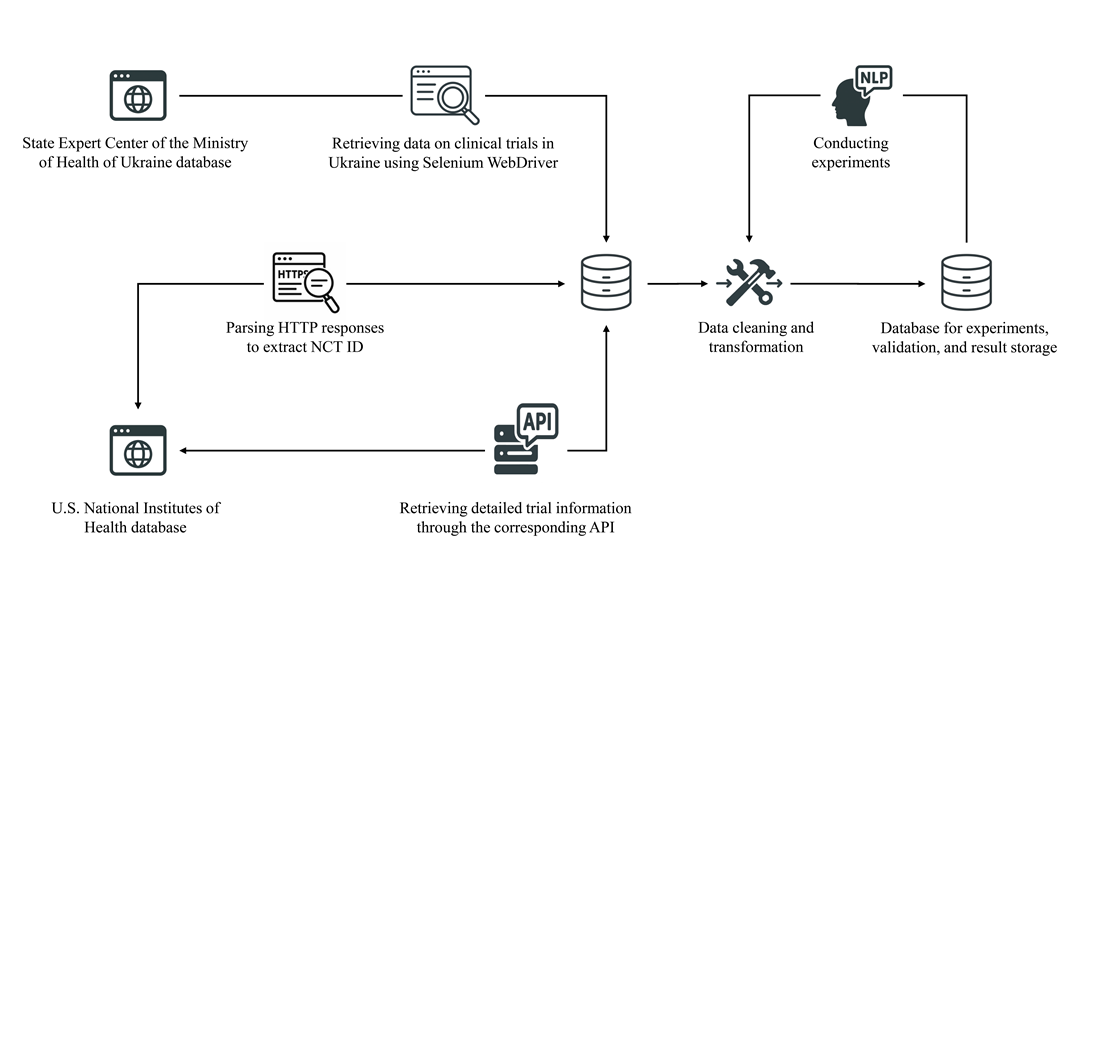
Об’єктом дослідження є неструктуровані текстові дані про клінічні випробування. Проблема полягає в тому, що аналіз таких даних є трудомістким і схильним до помилок процесом, навіть для фахівців. У свою чергу, це призводить до збільшення тривалості досліджень та затримок із випуском новітніх препаратів на ринок. У даній роботі продемонстровано підхід до формування набору даних про клінічні випробування, а також подальшого видобування ключової інформації за допомогою сучасних великих мовних моделей. Проведено дослідження з видобування таких показників, як допустима стать учасників, фаза дослідження та профіль дослідження. Загалом було виконано 11 703 експериментів, у більшості з яких вдалося досягти високих результатів. Зокрема, середні значення при використанні моделі GPT-4o-mini були такими: F1-міра – 0,92; влучність – 0,98; повнота – 0,99; точність – 0,87. Видобування інформації з клінічної документації українською мовою продемонструвало схожі результати порівняно з англомовними аналогами. В окремих випадках, спостерігалася значна кількість хибно-позитивних результатів і показники були суттєво нижчими (найнижчі зафіксовані значення: F1-міра – 0,52; влучність – 0,82; повнота – 0,78; точність – 0,35). Для таких випадків було проаналізовано причини, сформульовано відповідні висновки та рекомендації. Крім того, результати експериментів допомогли виявити низку розбіжностей та помилок в офіційних реєстрах, що є яскравим прикладом практичного застосування. Іншими прикладами використання отриманого результату є можливість масштабування технології на додаткові типи даних, а також підтримка цифрової трансформації у медичній сфері. Такі результати складають передумови для автоматизації процесу клінічних випробування та пришвидшення виходу новітніх препаратів на ринок.
Посилання
- Cascini, F., Beccia, F., Causio, F. A., Melnyk, A., Zaino, A., Ricciardi, W. (2022). Scoping review of the current landscape of AI-based applications in clinical trials. Frontiers in Public Health, 10. https://doi.org/10.3389/fpubh.2022.949377
- Kreimeyer, K., Foster, M., Pandey, A., Arya, N., Halford, G., Jones, S. F. et al. (2017). Natural language processing systems for capturing and standardizing unstructured clinical information: A systematic review. Journal of Biomedical Informatics, 73, 14–29. https://doi.org/10.1016/j.jbi.2017.07.012
- Alexander, M., Solomon, B., Ball, D. L., Sheerin, M., Dankwa-Mullan, I., Preininger, A. M. et al. (2020). Evaluation of an artificial intelligence clinical trial matching system in Australian lung cancer patients. JAMIA Open, 3 (2), 209–215. https://doi.org/10.1093/jamiaopen/ooaa002
- Liu, X., Hersch, G. L., Khalil, I., Devarakonda, M. (2021). Clinical Trial Information Extraction with BERT. 2021 IEEE 9th International Conference on Healthcare Informatics (ICHI). IEEE, 505–506. https://doi.org/10.1109/ichi52183.2021.00092
- Li, J., Wei, Q., Ghiasvand, O., Chen, M., Lobanov, V., Weng, C., Xu, H. (2022). A comparative study of pre-trained language models for named entity recognition in clinical trial eligibility criteria from multiple corpora. BMC Medical Informatics and Decision Making, 22 (S3). https://doi.org/10.1186/s12911-022-01967-7
- Nori, H., Lee, Y. T., Zhang, S., Carignan, D., Edgar, R., Fusi, N. et al. (2023). Can generalist foundation models outcompete special-purpose tuning? Case study in medicine. arXiv preprint. arXiv:2311.16452. https://doi.org/10.48550/arXiv.2311.16452
- Lee, J.-M. (2024). Strategies for integrating ChatGPT and generative AI into clinical studies. Blood Research, 59 (1). https://doi.org/10.1007/s44313-024-00045-3
- Lee, K., Paek, H., Huang, L.-C., Hilton, C. B., Datta, S., Higashi, J. et al. (2024). SEETrials: Leveraging large language models for safety and efficacy extraction in oncology clinical trials. Informatics in Medicine Unlocked, 50, 101589. https://doi.org/10.1016/j.imu.2024.101589
- Wong, A., Plasek, J. M., Montecalvo, S. P., Zhou, L. (2018). Natural Language Processing and Its Implications for the Future of Medication Safety: A Narrative Review of Recent Advances and Challenges. Pharmacotherapy: The Journal of Human Pharmacology and Drug Therapy, 38 (8), 822–841. https://doi.org/10.1002/phar.2151
- Dyyak, I., Horlatch, V., Pasichnyk, T., Pasichnyk, V. (2024). Assessing generative pre-trained transformer 4 in clinical trial inclusion criteria matching. Proceedings of the CEUR Workshop, 3702, 305–316. Available at: https://ceur-ws.org/Vol-3702/paper25.pdf
- Kang, T., Zhang, S., Tang, Y., Hruby, G. W., Rusanov, A., Elhadad, N., Weng, C. (2017). EliIE: An open-source information extraction system for clinical trial eligibility criteria. Journal of the American Medical Informatics Association, 24 (6), 1062–1071. https://doi.org/10.1093/jamia/ocx019
- Zeng, K., Xu, Y., Lin, G., Liang, L., Hao, T. (2021). Automated classification of clinical trial eligibility criteria text based on ensemble learning and metric learning. BMC Medical Informatics and Decision Making, 21 (S2). https://doi.org/10.1186/s12911-021-01492-z
- Kury, F., Butler, A., Yuan, C., Fu, L., Sun, Y., Liu, H. et al. (2020). Chia, a large annotated corpus of clinical trial eligibility criteria. Scientific Data, 7 (1). https://doi.org/10.1038/s41597-020-00620-0
- Thirunavukarasu, A. J., Ting, D. S. J., Elangovan, K., Gutierrez, L., Tan, T. F., Ting, D. S. W. (2023). Large language models in medicine. Nature Medicine, 29 (8), 1930–1940. https://doi.org/10.1038/s41591-023-02448-8
- Veen, D. V., Uden, C. V., Blankemeier, L., Delbrouck, J.-B., Aali, A., Bluethgen, C. et al. (2023). Clinical Text Summarization: Adapting Large Language Models Can Outperform Human Experts. https://doi.org/10.21203/rs.3.rs-3483777/v1
- Luo, X., Chen, F., Zhu, D., Wang, L., Wang, Z., Liu, H. et al. (2024). Potential Roles of Large Language Models in the Production of Systematic Reviews and Meta-Analyses. Journal of Medical Internet Research, 26, e56780. https://doi.org/10.2196/56780
- Datta, S., Lee, K., Paek, H., Manion, F. J., Ofoegbu, N., Du, J. et al. (2023). AutoCriteria: a generalizable clinical trial eligibility criteria extraction system powered by large language models. Journal of the American Medical Informatics Association, 31 (2), 375–385. https://doi.org/10.1093/jamia/ocad218
- Kim, J., Quintana, Y. (2022). Review of the Performance Metrics for Natural Language Systems for Clinical Trials Matching. MEDINFO 2021: One World, One Health – Global Partnership for Digital Innovation. IOS Press Ebooks, 641–644. https://doi.org/10.3233/shti220156
- Jin, Q., Wang, Z., Floudas, C. S., Chen, F., Gong, C., Bracken-Clarke, D. et al. (2024). Matching patients to clinical trials with large language models. Nature Communications, 15 (1). https://doi.org/10.1038/s41467-024-53081-z
- Pasichnyk, V. (2025). Datasets of clinical trials in Ukraine. Figshare. Collection. https://doi.org/10.6084/m9.figshare.c.7887785.v1
- Pasichnyk, V. (2025). Results of key data extraction from clinical trial documentation. Figshare. Dataset. https://doi.org/10.6084/m9.figshare.29378450
##submission.downloads##
Опубліковано
Як цитувати
Номер
Розділ
Ліцензія
Авторське право (c) 2025 Vasyl Pasichnyk, Vitaliy Horlatch

Ця робота ліцензується відповідно до Creative Commons Attribution 4.0 International License.
Закріплення та умови передачі авторських прав (ідентифікація авторства) здійснюється у Ліцензійному договорі. Зокрема, автори залишають за собою право на авторство свого рукопису та передають журналу право першої публікації цієї роботи на умовах ліцензії Creative Commons CC BY. При цьому вони мають право укладати самостійно додаткові угоди, що стосуються неексклюзивного поширення роботи у тому вигляді, в якому вона була опублікована цим журналом, але за умови збереження посилання на першу публікацію статті в цьому журналі.
Ліцензійний договір – це документ, в якому автор гарантує, що володіє усіма авторськими правами на твір (рукопис, статтю, тощо).
Автори, підписуючи Ліцензійний договір з ПП «ТЕХНОЛОГІЧНИЙ ЦЕНТР», мають усі права на подальше використання свого твору за умови посилання на наше видання, в якому твір опублікований. Відповідно до умов Ліцензійного договору, Видавець ПП «ТЕХНОЛОГІЧНИЙ ЦЕНТР» не забирає ваші авторські права та отримує від авторів дозвіл на використання та розповсюдження публікації через світові наукові ресурси (власні електронні ресурси, наукометричні бази даних, репозитарії, бібліотеки тощо).
За відсутності підписаного Ліцензійного договору або за відсутністю вказаних в цьому договорі ідентифікаторів, що дають змогу ідентифікувати особу автора, редакція не має права працювати з рукописом.
Важливо пам’ятати, що існує і інший тип угоди між авторами та видавцями – коли авторські права передаються від авторів до видавця. В такому разі автори втрачають права власності на свій твір та не можуть його використовувати в будь-який спосіб.










