Реалізація моделей машинного навчання для визначення відповідної моделі передбачення функцій білків
DOI:
https://doi.org/10.15587/1729-4061.2022.263270Ключові слова:
передбачення функцій білків, класифікація, нейронні мережі, ProtCNN, двонаправлена довга короткострокова пам’ять (BiLSTM)Анотація
Передбачення функцій білків є важливою частиною анотації генома, яка може допомогти у вирішенні широкого спектру біологічних завдань. Існує безліч методів передбачення функцій білків. Однак, за винятком послідовності, більшість ознак важко отримати або вони недоступні для багатьох білків, що обмежує область їхнього застосування. Крім того, ефективність методів передбачення ознак на основі послідовностей часто нижча, ніж у методів, що включають декілька ознак, а передбачення ознак білків може займати багато часу. Останні досягнення в цій області пов’язані з розвитком машинного навчання, що демонструє великий прогрес у вирішенні задачі передбачення функцій білків. Однак сьогодні більшість білкових послідовностей мають статус «нехарактерних» або «припустимих».
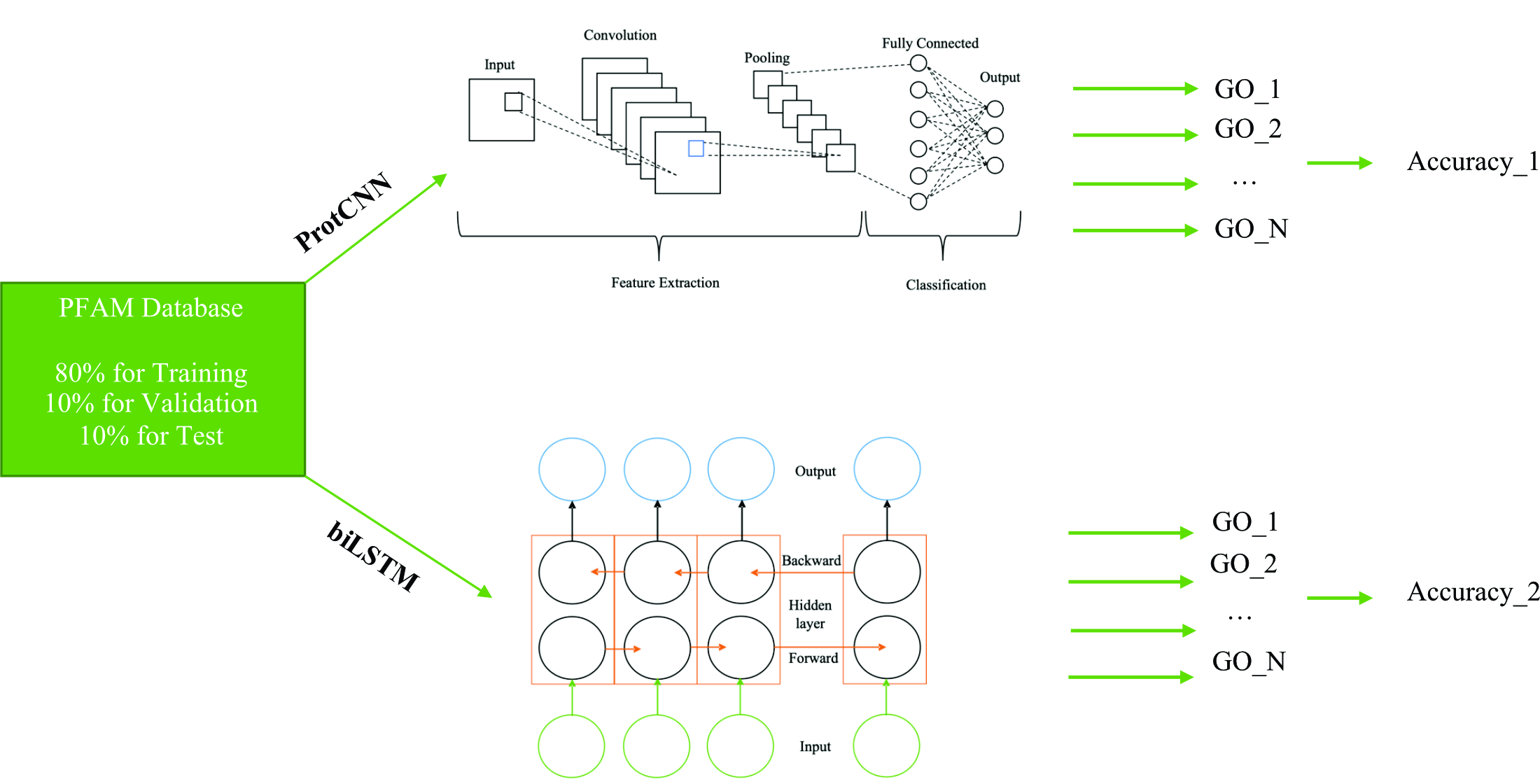
Необхідність оцінки точності ідентифікації функцій білків є актуальним завданням методів машинного навчання, що використовуються для передбачення функцій білків. В даному дослідженні оцінювалася ефективність двох популярних алгоритмів передбачення функцій (ProtCNN і BiLSTM) з двох точок зору, та були описані процедури побудови цих моделей.
В результаті дослідження сімейств Pfam, ProtCNN досягає точності 0,988 %, а двонаправлена LSTM має точність 0,9506 %. Використання набору даних Pfam дозволило підвищити точність класифікації за рахунок великого набору навчальних даних. При великому обсязі навчальних даних якість передбачення збільшується.
Дослідження показало, що алгоритми машинного навчання можуть використовуватися в якості ефективного інструменту для побудови моделей прогнозування функцій білків, зокрема, мережа CNN може бути адаптована в якості точного інструменту для анотації функцій білків за наявності великих наборів даних.
Посилання
- Gabaldon, T., Huynen, M. A. (2004). Prediction of protein function and pathways in the genome era. Cellular and Molecular Life Sciences (CMLS), 61 (7-8), 930–944. doi: https://doi.org/10.1007/s00018-003-3387-y
- du Plessis, L., Skunca, N., Dessimoz, C. (2011). The what, where, how and why of gene ontology--a primer for bioinformaticians. Briefings in Bioinformatics, 12 (6), 723–735. doi: https://doi.org/10.1093/bib/bbr002
- Barrell, D., Dimmer, E., Huntley, R. P., Binns, D., O’Donovan, C., Apweiler, R. (2009). The GOA database in 2009--an integrated Gene Ontology Annotation resource. Nucleic Acids Research, 37, D396–D403. doi: https://doi.org/10.1093/nar/gkn803
- Piovesan, D., Giollo, M., Leonardi, E., Ferrari, C., Tosatto, S. C. E. (2015). INGA: protein function prediction combining interaction networks, domain assignments and sequence similarity. Nucleic Acids Research, 43 (W1), W134–W140. doi: https://doi.org/10.1093/nar/gkv523
- Boratyn, G. M., Camacho, C., Cooper, P. S., Coulouris, G., Fong, A., Ma, N. et. al. (2013). BLAST: a more efficient report with usability improvements. Nucleic Acids Research, 41 (W1), W29–W33. doi: https://doi.org/10.1093/nar/gkt282
- Stephenson, N., Shane, E., Chase, J., Rowland, J., Ries, D., Justice, N. et. al. (2019). Survey of Machine Learning Techniques in Drug Discovery. Current Drug Metabolism, 20 (3), 185–193. doi: https://doi.org/10.2174/1389200219666180820112457
- Lobley, A. E., Nugent, T., Orengo, C. A., Jones, D. T. (2008). FFPred: an integrated feature-based function prediction server for vertebrate proteomes. Nucleic Acids Research, 36, W297–W302. doi: https://doi.org/10.1093/nar/gkn193
- Cozzetto, D., Minneci, F., Currant, H., Jones, D. T. (2016). FFPred 3: feature-based function prediction for all Gene Ontology domains. Scientific Reports, 6 (1). doi: https://doi.org/10.1038/srep31865
- Jung, J., Yi, G., Sukno, S. A., Thon, M. R. (2010). PoGO: Prediction of Gene Ontology terms for fungal proteins. BMC Bioinformatics, 11 (1). doi: https://doi.org/10.1186/1471-2105-11-215
- Törönen, P., Medlar, A., Holm, L. (2018). PANNZER2: a rapid functional annotation web server. Nucleic Acids Research, 46 (W1), W84–W88. doi: https://doi.org/10.1093/nar/gky350
- You, R., Huang, X., Zhu, S. (2018). DeepText2GO: Improving large-scale protein function prediction with deep semantic text representation. Methods, 145, 82–90. doi: https://doi.org/10.1016/j.ymeth.2018.05.026
- You, R., Yao, S., Xiong, Y., Huang, X., Sun, F., Mamitsuka, H., Zhu, S. (2019). NetGO: improving large-scale protein function prediction with massive network information. Nucleic Acids Research, 47 (W1), W379–W387. doi: https://doi.org/10.1093/nar/gkz388
- Kulmanov, M., Khan, M. A., Hoehndorf, R. (2017). DeepGO: predicting protein functions from sequence and interactions using a deep ontology-aware classifier. Bioinformatics, 34 (4), 660–668. doi: https://doi.org/10.1093/bioinformatics/btx624
- Cai, Y., Wang, J., Deng, L. (2020). SDN2GO: An Integrated Deep Learning Model for Protein Function Prediction. Frontiers in Bioengineering and Biotechnology, 8. doi: https://doi.org/10.3389/fbioe.2020.00391
- Du, Z., He, Y., Li, J., Uversky, V. N. (2020). DeepAdd: Protein function prediction from k-mer embedding and additional features. Computational Biology and Chemistry, 89, 107379. doi: https://doi.org/10.1016/j.compbiolchem.2020.107379
- Zhang, F., Song, H., Zeng, M., Wu, F.-X., Li, Y., Pan, Y., Li, M. (2021). A Deep Learning Framework for Gene Ontology Annotations With Sequence- and Network-Based Information. IEEE/ACM Transactions on Computational Biology and Bioinformatics, 18 (6), 2208–2217. doi: https://doi.org/10.1109/tcbb.2020.2968882
- Spalević, S., Veličković, P., Kovačević, J., Nikolić, M. (2020). Hierarchical Protein Function Prediction with Tail-GNNs. arXiv. doi: https://doi.org/10.48550/arXiv.2007.12804
- LeCun, Y., Bengio, Y., Hinton, G. (2015). Deep learning. Nature, 521 (7553), 436–444. doi: https://doi.org/10.1038/nature14539
- Cao, R., Freitas, C., Chan, L., Sun, M., Jiang, H., Chen, Z. (2017). ProLanGO: Protein Function Prediction Using Neural Machine Translation Based on a Recurrent Neural Network. Molecules, 22 (10), 1732. doi: https://doi.org/10.3390/molecules22101732
- Jiang, Y., Oron, T. R., Clark, W. T., Bankapur, A. R., D’Andrea, D., Lepore, R. et. al. (2016). An expanded evaluation of protein function prediction methods shows an improvement in accuracy. Genome Biology, 17 (1). doi: https://doi.org/10.1186/s13059-016-1037-6
- Pearson, W. R. (2015). Protein Function Prediction: Problems and Pitfalls. Current Protocols in Bioinformatics, 51 (1). doi: https://doi.org/10.1002/0471250953.bi0412s51
- UniProt: the universal protein knowledgebase (2016). Nucleic Acids Research, 45 (D1), D158–D169. doi: https://doi.org/10.1093/nar/gkw1099
- Pfam 35.0 is released. Xfam Blog. Available at: https://xfam.wordpress.com/2021/11/19/pfam-35-0-is-released/
- Bileschi, M. L., Belanger, D., Bryant, D., Sanderson, T., Carter, B., Sculley, D. et. al. (2019). Using Deep Learning to Annotate the Protein Universe. bioRxiv. doi: https://doi.org/10.1101/626507
- Vu, T. T. D., Jung, J. (2021). Protein function prediction with gene ontology: from traditional to deep learning models. PeerJ, 9, e12019. doi: https://doi.org/10.7717/peerj.12019
- Abduljabbar, R. L., Dia, H., Tsai, P.-W. (2021). Unidirectional and Bidirectional LSTM Models for Short-Term Traffic Prediction. Journal of Advanced Transportation, 2021, 1–16. doi: https://doi.org/10.1155/2021/5589075
- Kurtukova, A. V., Romanov, A. S. (2019). Modeling the neural network architecture to identify the author of the source code. Proceedings of Tomsk State University of Control Systems and Radioelectronics, 22 (3), 37–42. doi: https://doi.org/10.21293/1818-0442-2019-22-3-37-42
- Deen, A., Gayanchandani, M. (2019). Protein Function Prediction using SVM Kernel Approach. International Journal of Scientific & Engineering Research, 10 (7), 1995–2000. Available at: https://www.ijser.org/researchpaper/Protein-Function-Prediction-using-SVM-Kernel-Approach.pdf
- Kingma, D. P., Ba, J. (2014). Adam: A Method for Stochastic Optimization. 3rd International Conference for Learning Representations. San Diego. doi: https://doi.org/10.48550/arXiv.1412.6980
##submission.downloads##
Опубліковано
Як цитувати
Номер
Розділ
Ліцензія
Авторське право (c) 2022 Yekaterina Golenko, Aisulu Ismailova, Anargul Shaushenova, Zhazira Mutalova, Damir Dossalyanov, Aliya Ainagulova, Akgul Naizagarayeva

Ця робота ліцензується відповідно до Creative Commons Attribution 4.0 International License.
Закріплення та умови передачі авторських прав (ідентифікація авторства) здійснюється у Ліцензійному договорі. Зокрема, автори залишають за собою право на авторство свого рукопису та передають журналу право першої публікації цієї роботи на умовах ліцензії Creative Commons CC BY. При цьому вони мають право укладати самостійно додаткові угоди, що стосуються неексклюзивного поширення роботи у тому вигляді, в якому вона була опублікована цим журналом, але за умови збереження посилання на першу публікацію статті в цьому журналі.
Ліцензійний договір – це документ, в якому автор гарантує, що володіє усіма авторськими правами на твір (рукопис, статтю, тощо).
Автори, підписуючи Ліцензійний договір з ПП «ТЕХНОЛОГІЧНИЙ ЦЕНТР», мають усі права на подальше використання свого твору за умови посилання на наше видання, в якому твір опублікований. Відповідно до умов Ліцензійного договору, Видавець ПП «ТЕХНОЛОГІЧНИЙ ЦЕНТР» не забирає ваші авторські права та отримує від авторів дозвіл на використання та розповсюдження публікації через світові наукові ресурси (власні електронні ресурси, наукометричні бази даних, репозитарії, бібліотеки тощо).
За відсутності підписаного Ліцензійного договору або за відсутністю вказаних в цьому договорі ідентифікаторів, що дають змогу ідентифікувати особу автора, редакція не має права працювати з рукописом.
Важливо пам’ятати, що існує і інший тип угоди між авторами та видавцями – коли авторські права передаються від авторів до видавця. В такому разі автори втрачають права власності на свій твір та не можуть його використовувати в будь-який спосіб.









