Розробка згорткової нейронної мережі для класифікації зображень пухлин за допомогою Inception v3
DOI:
https://doi.org/10.15587/1729-4061.2023.281227Ключові слова:
згорткова нейронна мережа, глибоке навчання, класифікація, архітектура Inception, пухлина головного мозкуАнотація
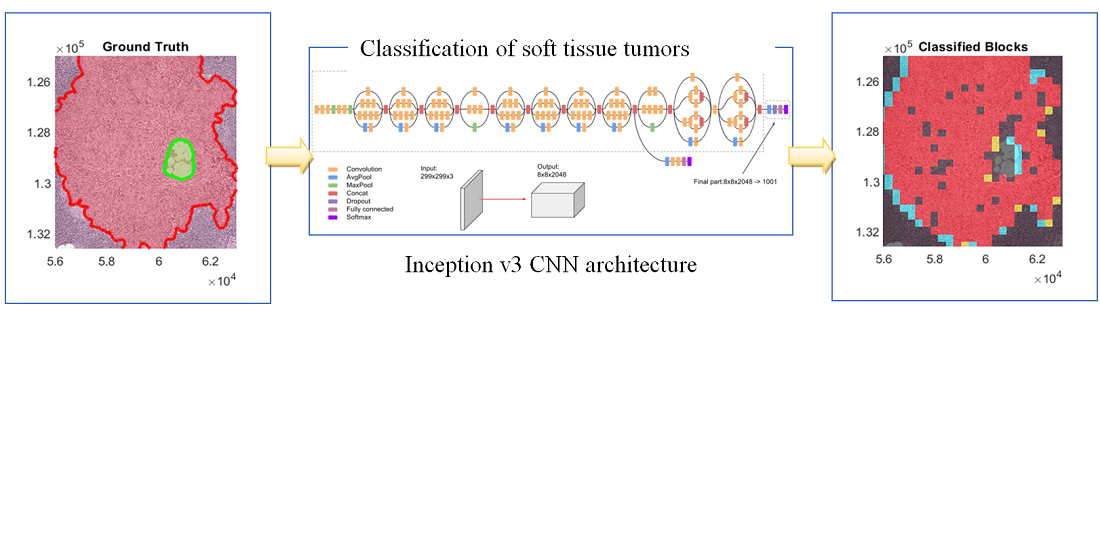
Алгоритми глибокого навчання спираються на цифрову патологію для класифікації пухлин тканин, коли цілі слайди тканин оцифровуються та візуалізуються. Отримані повнослайдові зображення зі змінною роздільною здатністю (MWSI) мають високу роздільну здатність, яка може коливатися приблизно від 100 000 до 200 000 пікселів. Для спрощення обробки зображень, навігації та ефективної експозиції MWSI часто зберігаються в конфігурації зі змінною роздільною здатністю. У даній роботі з використанням глибокої нейронної архітектури Inception-v3 розроблена мережа для класифікації MWSI, що потребують великого обсягу пам'яті. У роботі використовуються MWSI з Camelyon16, розмір якого становить близько 451 ГБ набору даних Challenge з двох незалежних джерел, включаючи 400 MWSI для лімфатичних вузлів. Навчальний набір даних містить 111 MWSI пухлинної тканини і лімфатичних вузлів та 159 WSI нормальних лімфатичних вузлів. У розробленій моделі використовується обробка на основі зразків для навчання великих MWSI за допомогою платформи MATLAB. У моделі представлені методи трансферного навчання з архітектурою на основі Inception-v3 для класифікації окремих зразків як пухлини або норми. Таким чином, основною метою є досягнення бінарної сегментації за двома класами, включаючи норму та пухлину. Це передбачає створення нового повнозв'язаного шару для архітектури Inception-v3 з двома класами та компенсацію нових шарів замість вихідних кінцевих повнозв'язних шарів. Отримані результати показали, що візуалізація теплової карти дозволяє розпізнавати граничні координати основної істини як схематичну область інтересу (ROI), де зелена межа представляє нормальні області, а червоні межі - область пухлини. Запропонована архітектура згорткової нейронної мережі (ЗНМ) Inception v3 дозволяє досягти точності понад 92,8 % для такого набору даних MWSI для класифікації пухлин головного мозку на нормальну та пухлинну тканини
Посилання
- Abdelaziz Ismael, S. A., Mohammed, A., Hefny, H. (2020). An enhanced deep learning approach for brain cancer MRI images classification using residual networks. Artificial Intelligence in Medicine, 102, 101779. doi: https://doi.org/10.1016/j.artmed.2019.101779
- Sfayyih, A. H., Sabry, A. H., Jameel, S. M., Sulaiman, N., Raafat, S. M., Humaidi, A. J., Kubaiaisi, Y. M. A. (2023). Acoustic-Based Deep Learning Architectures for Lung Disease Diagnosis: A Comprehensive Overview. Diagnostics, 13 (10), 1748. doi: https://doi.org/10.3390/diagnostics13101748
- Abd-Ellah, M. K., Awad, A. I., Khalaf, A. A. M., Hamed, H. F. A. (2018). Two-phase multi-model automatic brain tumour diagnosis system from magnetic resonance images using convolutional neural networks. EURASIP Journal on Image and Video Processing, 2018 (1). doi: https://doi.org/10.1186/s13640-018-0332-4
- Abd El Kader, I., Xu, G., Shuai, Z., Saminu, S., Javaid, I., Salim Ahmad, I. (2021). Differential Deep Convolutional Neural Network Model for Brain Tumor Classification. Brain Sciences, 11 (3), 352. doi: https://doi.org/10.3390/brainsci11030352
- Houssein, E. H., Emam, M. M., Ali, A. A., Suganthan, P. N. (2021). Deep and machine learning techniques for medical imaging-based breast cancer: A comprehensive review. Expert Systems with Applications, 167, 114161. doi: https://doi.org/10.1016/j.eswa.2020.114161
- Zhen, S., Cheng, M., Tao, Y., Wang, Y., Juengpanich, S., Jiang, Z. et al. (2020). Deep Learning for Accurate Diagnosis of Liver Tumor Based on Magnetic Resonance Imaging and Clinical Data. Frontiers in Oncology, 10. doi: https://doi.org/10.3389/fonc.2020.00680
- Alqudah, A. M. (2019). Brain Tumor Classification Using Deep Learning Technique - A Comparison between Cropped, Uncropped, and Segmented Lesion Images with Different Sizes. International Journal of Advanced Trends in Computer Science and Engineering, 8 (6), 3684–3691. doi: https://doi.org/10.30534/ijatcse/2019/155862019
- Wang, H., Zhou, Z., Li, Y., Chen, Z., Lu, P., Wang, W. et al. (2017). Comparison of machine learning methods for classifying mediastinal lymph node metastasis of non-small cell lung cancer from 18F-FDG PET/CT images. EJNMMI Research, 7 (1). doi: https://doi.org/10.1186/s13550-017-0260-9
- Hoseini, F., Shahbahrami, A., Bayat, P. (2018). An Efficient Implementation of Deep Convolutional Neural Networks for MRI Segmentation. Journal of Digital Imaging, 31 (5), 738–747. doi: https://doi.org/10.1007/s10278-018-0062-2
- Wei, J. W., Tafe, L. J., Linnik, Y. A., Vaickus, L. J., Tomita, N., Hassanpour, S. (2019). Pathologist-level classification of histologic patterns on resected lung adenocarcinoma slides with deep neural networks. Scientific Reports, 9 (1). doi: https://doi.org/10.1038/s41598-019-40041-7
- Mehrotra, R., Ansari, M. A., Agrawal, R., Anand, R. S. (2020). A Transfer Learning approach for AI-based classification of brain tumors. Machine Learning with Applications, 2, 100003. doi: https://doi.org/10.1016/j.mlwa.2020.100003
- Advanced Guide to Inception v3. Cloud TPU. Google Cloud. Available at: https://cloud.google.com/tpu/docs/inception-v3-advanced
- Litjens, G., Bandi, P., Ehteshami Bejnordi, B., Geessink, O., Balkenhol, M., Bult, P. et al. (2018). 1399 H&E-stained sentinel lymph node sections of breast cancer patients: the CAMELYON dataset. GigaScience, 7 (6). doi: https://doi.org/10.1093/gigascience/giy065
- Szegedy, C., Vanhoucke, V., Ioffe, S., Shlens, J., Wojna, Z. (2016). Rethinking the Inception Architecture for Computer Vision. 2016 IEEE Conference on Computer Vision and Pattern Recognition (CVPR). doi: https://doi.org/10.1109/cvpr.2016.308
- ImageNet. Available at:https://www.image-net.org/
- Raharjo, B., Farida, N., Subekti, P., Herlina, S., Doddy, H., Rahim, R. (2021). Optimization forecasting using back-propagation algorithm. Journal of Applied Engineering Science, 19 (4), 1083–1089. doi: https://doi.org/10.5937/jaes0-30175
- Negassi, M., Suarez-Ibarrola, R., Hein, S., Miernik, A., Reiterer, A. (2020). Application of artificial neural networks for automated analysis of cystoscopic images: a review of the current status and future prospects. World Journal of Urology, 38 (10), 2349–2358. doi: https://doi.org/10.1007/s00345-019-03059-0
- Zhuang, Z., Ding, W., Zhuang, S., Joseph Raj, A. N., Wang, J., Zhou, W., Wei, C. (2021). Tumor classification in automated breast ultrasound (ABUS) based on a modified extracting feature network. Computerized Medical Imaging and Graphics, 90, 101925. doi: https://doi.org/10.1016/j.compmedimag.2021.101925
- Alanazi, M. F., Ali, M. U., Hussain, S. J., Zafar, A., Mohatram, M., Irfan, M., AlRuwaili, R. et al. (2022). Brain Tumor/Mass Classification Framework Using Magnetic-Resonance-Imaging-Based Isolated and Developed Transfer Deep-Learning Model. Sensors, 22 (1), 372. doi: https://doi.org/10.3390/s22010372
- Mahmood, T., Li, J., Pei, Y., Akhtar, F., Rehman, M. U., Wasti, S. H. (2022). Breast lesions classifications of mammographic images using a deep convolutional neural network-based approach. PLOS ONE, 17 (1), e0263126. doi: https://doi.org/10.1371/journal.pone.0263126
- Asif, S., Yi, W., Ain, Q. U., Hou, J., Yi, T., Si, J. (2022). Improving Effectiveness of Different Deep Transfer Learning-Based Models for Detecting Brain Tumors From MR Images. IEEE Access, 10, 34716–34730. doi: https://doi.org/10.1109/access.2022.3153306
- Jwaid, W. M., Al-Husseini, Z. S. M., Sabry, A. H. (2021). Development of brain tumor segmentation of magnetic resonance imaging (MRI) using U-Net deep learning. Eastern-European Journal of Enterprise Technologies, 4 (9 (112)), 23–31. doi: https://doi.org/10.15587/1729-4061.2021.238957
##submission.downloads##
Опубліковано
Як цитувати
Номер
Розділ
Ліцензія
Авторське право (c) 2023 Ali A. Mahmood, Sadeer Sadeq, Yaser Issam Aljanabi, Ahmad H. Sabry

Ця робота ліцензується відповідно до Creative Commons Attribution 4.0 International License.
Закріплення та умови передачі авторських прав (ідентифікація авторства) здійснюється у Ліцензійному договорі. Зокрема, автори залишають за собою право на авторство свого рукопису та передають журналу право першої публікації цієї роботи на умовах ліцензії Creative Commons CC BY. При цьому вони мають право укладати самостійно додаткові угоди, що стосуються неексклюзивного поширення роботи у тому вигляді, в якому вона була опублікована цим журналом, але за умови збереження посилання на першу публікацію статті в цьому журналі.
Ліцензійний договір – це документ, в якому автор гарантує, що володіє усіма авторськими правами на твір (рукопис, статтю, тощо).
Автори, підписуючи Ліцензійний договір з ПП «ТЕХНОЛОГІЧНИЙ ЦЕНТР», мають усі права на подальше використання свого твору за умови посилання на наше видання, в якому твір опублікований. Відповідно до умов Ліцензійного договору, Видавець ПП «ТЕХНОЛОГІЧНИЙ ЦЕНТР» не забирає ваші авторські права та отримує від авторів дозвіл на використання та розповсюдження публікації через світові наукові ресурси (власні електронні ресурси, наукометричні бази даних, репозитарії, бібліотеки тощо).
За відсутності підписаного Ліцензійного договору або за відсутністю вказаних в цьому договорі ідентифікаторів, що дають змогу ідентифікувати особу автора, редакція не має права працювати з рукописом.
Важливо пам’ятати, що існує і інший тип угоди між авторами та видавцями – коли авторські права передаються від авторів до видавця. В такому разі автори втрачають права власності на свій твір та не можуть його використовувати в будь-який спосіб.









