Порівняльна оцінка моделей на основі unet із підсиленням шуму для сегментації раку молочної залози на ультразвукових зображеннях
DOI:
https://doi.org/10.15587/1729-4061.2023.289044Ключові слова:
рак молочної залози, ультразвукове зображення, глибоке навчання, UNet, сегментація зображення, збільшенняАнотація
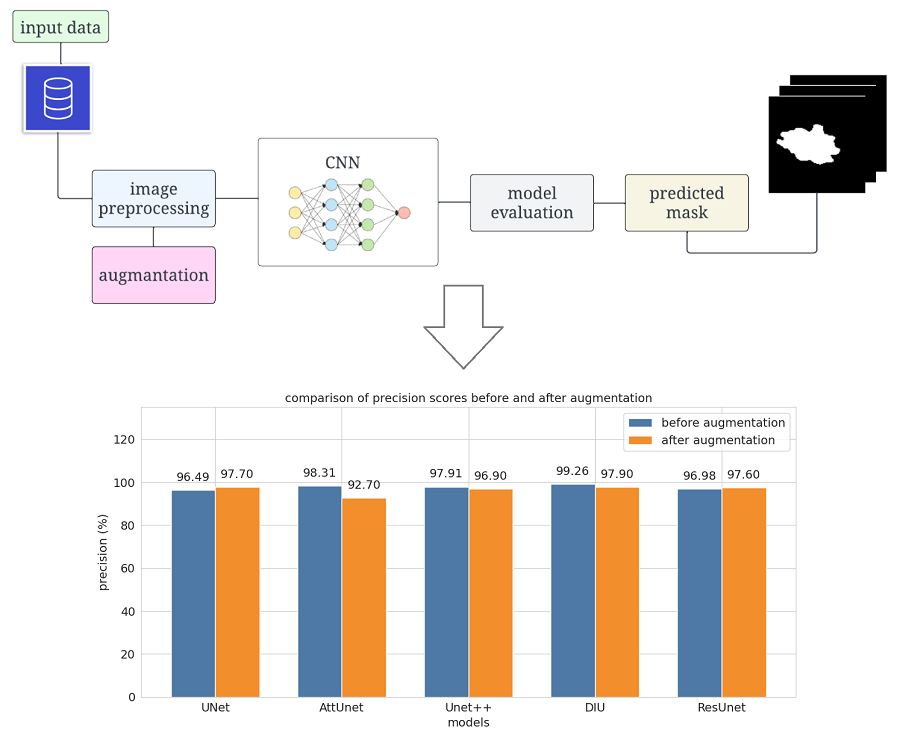
У цій статті досліджується застосування згорткових нейронних мереж (ЗНМ), зокрема архітектури моделі UNet, для підвищення точності сегментації пухлини раку молочної залози на ультразвукових зображеннях. Точна ідентифікація раку молочної залози є важливою для ефективного лікування пацієнта. Однак ультразвукові зображення часто містять шуми та артефакти, що може ускладнити завдання сегментації пухлини. Тому, щоб підкреслити найбільш надійну архітектуру, до оригінального набору було внесено зміни, зокрема додано шум і нечіткість. У цьому дослідженні було проведено порівняльне дослідження п’яти різних варіантів моделей UNet (UNet, Attention UNet, UNet++, Dense Inception UNet і Residual UNet) на різноманітних ультразвукових зображеннях різних пухлин молочної залози. Використовуючи послідовні методи навчання та прийоми розширення та додавання шуму до даних, було підкреслено покращення точності сегментації при використанні архітектури Dense Inception UNet. Результати мають потенційне практичне застосування в галузі медичної діагностики та можуть допомогти медичним працівникам у автоматичній сегментації пухлини на ультразвукових зображеннях раку молочної залози. Це дослідження підкреслює підвищення точності сегментації шляхом впровадження щільної індукції в архітектуру UNet. Важливо, що коефіцієнт Дайса, ключовий показник сегментації, помітно покращився, збільшившись із 0,973 до 0,976 після доповнення даних. Результати дослідження є перспективними для медичної спільноти, пропонуючи більш точний і надійний підхід до сегментації уражень раку молочної залози на ультразвукових зображеннях. Отримані результати можуть бути використані в клінічній практиці, щоб допомогти радіологам у ранній діагностиці раку
Посилання
- World Health Organization. Available at: https://www.who.int/
- Dunenova, G. A., Kalmataeva, Z. A., Kaidarova, D. R., Shatkovskaya, O. V., Zhylkaidarova, A. Z., Marchenko, E. A.,Glushkova, N. E. (2023). Breast cancer epidemiology in Kazakhstan for the period 2012-2021. Science & Healthcare, 25 (2). doi: https://doi.org/10.34689/SH.2023.25.2.018
- Igissinov, N., Toguzbayeva, A., Turdaliyeva, B., Igissinova, G., Bilyalova, Z., Akpolatova, G. et al. (2020). Breast Cancer in Megapolises of Kazakhstan: Epidemiological Assessment of Incidence and Mortality. Iranian Journal of Public Health. doi: https://doi.org/10.18502/ijph.v48i7.2948
- Barba, D., León-Sosa, A., Lugo, P., Suquillo, D., Torres, F., Surre, F. et al. (2021). Breast cancer, screening and diagnostic tools: All you need to know. Critical Reviews in Oncology/Hematology, 157, 103174. doi: https://doi.org/10.1016/j.critrevonc.2020.103174
- Ginsburg, O., Yip, C., Brooks, A., Cabanes, A., Caleffi, M., Dunstan Yataco, J. A. et al. (2020). Breast cancer early detection: A phased approach to implementation. Cancer, 126 (S10), 2379–2393. doi: https://doi.org/10.1002/cncr.32887
- Esteva, A., Chou, K., Yeung, S., Naik, N., Madani, A., Mottaghi, A. et al. (2021). Deep learning-enabled medical computer vision. Npj Digital Medicine, 4 (1). doi: https://doi.org/10.1038/s41746-020-00376-2
- Dhahri, H., Al Maghayreh, E., Mahmood, A., Elkilani, W., Faisal Nagi, M. (2019). Automated Breast Cancer Diagnosis Based on Machine Learning Algorithms. Journal of Healthcare Engineering, 2019, 1–11. doi: https://doi.org/10.1155/2019/4253641
- MI-STA 2022 Conference Proceeding. (2022). 2022 IEEE 2nd International Maghreb Meeting of the Conference on Sciences and Techniques of Automatic Control and Computer Engineering (MI-STA). doi: https://doi.org/10.1109/mi-sta54861.2022.9837515
- Kuttan, G. O., Elayidom, M. S. (2023). Review on Computer Aided Breast Cancer Detection and Diagnosis using Machine Learning Methods on Mammogram Image. Current Medical Imaging Formerly Current Medical Imaging Reviews, 19 (12). doi: https://doi.org/10.2174/1573405619666230213093639
- Liu, Z., Wang, S., Dong, D., Wei, J., Fang, C., Zhou, X. et al. (2019). The Applications of Radiomics in Precision Diagnosis and Treatment of Oncology: Opportunities and Challenges. Theranostics, 9 (5), 1303–1322. doi: https://doi.org/10.7150/thno.30309
- Ronneberger, O., Fischer, P., Brox, T. (2015). U-Net: Convolutional Networks for Biomedical Image Segmentation. Medical Image Computing and Computer-Assisted Intervention – MICCAI 2015, 234–241. doi: https://doi.org/10.1007/978-3-319-24574-4_28
- Zhou, Z., Rahman Siddiquee, M. M., Tajbakhsh, N., Liang, J. (2018). UNet++: A Nested U-Net Architecture for Medical Image Segmentation. Lecture Notes in Computer Science, 3–11. doi: https://doi.org/10.1007/978-3-030-00889-5_1
- Oktay, O., Schlemper, J., Folgoc, L. L., Lee, M., Heinrich, M., Misawa, K. et al. (2018). Attention u-net: Learning where to look for the pancreas. arXiv. doi: https://doi.org/10.48550/arXiv.1804.03999
- Zhang, Z., Liu, Q., Wang, Y. (2018). Road Extraction by Deep Residual U-Net. IEEE Geoscience and Remote Sensing Letters, 15 (5), 749–753. doi: https://doi.org/10.1109/lgrs.2018.2802944
- Targ, S., Almeida, D., Lyman, K. (2016). Resnet in resnet: Generalizing residual architectures. arXiv. doi: https://doi.org/10.48550/arXiv.1603.08029
- Zhang, Z., Wu, C., Coleman, S., Kerr, D. (2020). DENSE-INception U-net for medical image segmentation. Computer Methods and Programs in Biomedicine, 192, 105395. doi: https://doi.org/10.1016/j.cmpb.2020.105395
- Zhao, Y., Lai, Z., Shen, L., Kong, H. (2022). Breast Lesions Segmentation using Dual-level UNet (DL-UNet). 2022 IEEE 35th International Symposium on Computer-Based Medical Systems (CBMS). doi: https://doi.org/10.1109/cbms55023.2022.00067
- Honghan, Z., Liu, D. C., Jingyan, L., Liu, P., Yin, H., Peng, Y. (2021). RMS-SE-UNet: A Segmentation Method for Tumors in Breast Ultrasound Images. 2021 IEEE 6th International Conference on Computer and Communication Systems (ICCCS). doi: https://doi.org/10.1109/icccs52626.2021.9449302
- Negi, A., Raj, A. N. J., Nersisson, R., Zhuang, Z., Murugappan, M. (2020). RDA-UNET-WGAN: An Accurate Breast Ultrasound Lesion Segmentation Using Wasserstein Generative Adversarial Networks. Arabian Journal for Science and Engineering, 45 (8), 6399–6410. doi: https://doi.org/10.1007/s13369-020-04480-z
- Chavan, T., Prajapati, K., JV, K. R. (2022). InvUNET: Involuted UNET for Breast Tumor Segmentation from Ultrasound. Lecture Notes in Computer Science, 283–290. doi: https://doi.org/10.1007/978-3-031-09342-5_27
- Chen, G., Li, L., Dai, Y., Zhang, J., Yap, M. H. (2023). AAU-Net: An Adaptive Attention U-Net for Breast Lesions Segmentation in Ultrasound Images. IEEE Transactions on Medical Imaging, 42 (5), 1289–1300. doi: https://doi.org/10.1109/tmi.2022.3226268
- Agarap, A. F. (2018). Deep Learning using Rectified Linear Units (ReLU). arXiv. doi: https://doi.org/10.48550/arXiv.1803.08375
- Liang, X., Wang, X., Lei, Z., Liao, S., Li, S. Z. (2017). Soft-Margin Softmax for Deep Classification. Lecture Notes in Computer Science, 413–421. doi: https://doi.org/10.1007/978-3-319-70096-0_43
- Guerrero-Pena, F. A., Marrero Fernandez, P. D., Ing Ren, T., Yui, M., Rothenberg, E., Cunha, A. (2018). Multiclass Weighted Loss for Instance Segmentation of Cluttered Cells. 2018 25th IEEE International Conference on Image Processing (ICIP). doi: https://doi.org/10.1109/icip.2018.8451187
- Schlemper, J., Oktay, O., Schaap, M., Heinrich, M., Kainz, B., Glocker, B., Rueckert, D. (2019). Attention gated networks: Learning to leverage salient regions in medical images. Medical Image Analysis, 53, 197–207. doi: https://doi.org/10.1016/j.media.2019.01.012
- Khanna, A., Londhe, N. D., Gupta, S., Semwal, A. (2020). A deep Residual U-Net convolutional neural network for automated lung segmentation in computed tomography images. Biocybernetics and Biomedical Engineering, 40 (3), 1314–1327. doi: https://doi.org/10.1016/j.bbe.2020.07.007
- He, K., Zhang, X., Ren, S., Sun, J. (2016). Deep Residual Learning for Image Recognition. 2016 IEEE Conference on Computer Vision and Pattern Recognition (CVPR). doi: https://doi.org/10.1109/cvpr.2016.90
- Chicco, D., Jurman, G. (2020). The advantages of the Matthews correlation coefficient (MCC) over F1 score and accuracy in binary classification evaluation. BMC Genomics, 21 (1). doi: https://doi.org/10.1186/s12864-019-6413-7
- Shamir, R. R., Duchin, Y., Kim, J., Sapiro, G., Harel, N. (2018). Continuous Dice Coefficient: a Method for Evaluating Probabilistic Segmentations. doi: https://doi.org/10.1101/306977
- Derczynski, L. (2016). Complementarity, F-score, and NLP Evaluation. Proceedings of the Tenth International Conference on Language Resources and Evaluation (LREC'16). Portorož, 261–266. Available at: https://aclanthology.org/L16-1040/
- Eelbode, T., Bertels, J., Berman, M., Vandermeulen, D., Maes, F., Bisschops, R., Blaschko, M. B. (2020). Optimization for Medical Image Segmentation: Theory and Practice When Evaluating With Dice Score or Jaccard Index. IEEE Transactions on Medical Imaging, 39 (11), 3679–3690. doi: https://doi.org/10.1109/tmi.2020.3002417
- Cheng, B., Girshick, R., Dollar, P., Berg, A. C., Kirillov, A. (2021). Boundary IoU: Improving Object-Centric Image Segmentation Evaluation. 2021 IEEE/CVF Conference on Computer Vision and Pattern Recognition (CVPR). doi: https://doi.org/10.1109/cvpr46437.2021.01508
- Al-Dhabyani, W., Gomaa, M., Khaled, H., Fahmy, A. (2020). Dataset of breast ultrasound images. Data in Brief, 28, 104863. doi: https://doi.org/10.1016/j.dib.2019.104863
- Al-Dhabyani, W., Gomaa, M., Khaled, H., Fahmy, A. (2019). Deep Learning Approaches for Data Augmentation and Classification of Breast Masses using Ultrasound Images. International Journal of Advanced Computer Science and Applications, 10 (5). doi: https://doi.org/10.14569/ijacsa.2019.0100579
- Akkus, Z., Cai, J., Boonrod, A., Zeinoddini, A., Weston, A. D., Philbrick, K. A., Erickson, B. J. (2019). A Survey of Deep-Learning Applications in Ultrasound: Artificial Intelligence–Powered Ultrasound for Improving Clinical Workflow. Journal of the American College of Radiology, 16 (9), 1318–1328. doi: https://doi.org/10.1016/j.jacr.2019.06.004
- Ilesanmi, A. E., Chaumrattanakul, U., Makhanov, S. S. (2021). A method for segmentation of tumors in breast ultrasound images using the variant enhanced deep learning. Biocybernetics and Biomedical Engineering, 41 (2), 802–818. doi: https://doi.org/10.1016/j.bbe.2021.05.007
- Almajalid, R., Shan, J., Du, Y., Zhang, M. (2018). Development of a Deep-Learning-Based Method for Breast Ultrasound Image Segmentation. 2018 17th IEEE International Conference on Machine Learning and Applications (ICMLA). doi: https://doi.org/10.1109/icmla.2018.00179
- Zhang, C., Bao, N., Sun, H., Li, H., Li, J., Qian, W., Zhou, S. (2022). A Deep Learning Image Data Augmentation Method for Single Tumor Segmentation. Frontiers in Oncology, 12. doi: https://doi.org/10.3389/fonc.2022.782988
- Ferreira, M. R., Torres, H. R., Oliveira, B., Gomes-Fonseca, J., Morais, P., Novais, P., Vilaca, J. L. (2022). Comparative Analysis of Current Deep Learning Networks for Breast Lesion Segmentation in Ultrasound Images. 2022 44th Annual International Conference of the IEEE Engineering in Medicine & Biology Society (EMBC). doi: https://doi.org/10.1109/embc48229.2022.9871091
- Mukasheva, A., Akanov, Z., Yedilkhan, D. (2021). Research of the Regression Analysis Methods for Predicting the Growth of Patients with Diabetes Mellitus. 2021 IEEE International Conference on Smart Information Systems and Technologies (SIST). doi: https://doi.org/10.1109/sist50301.2021.9465975
- Zhao, T., Dai, H. (2022). Breast Tumor Ultrasound Image Segmentation Method Based on Improved Residual U-Net Network. Computational Intelligence and Neuroscience, 2022, 1–9. doi: https://doi.org/10.1155/2022/3905998
##submission.downloads##
Опубліковано
Як цитувати
Номер
Розділ
Ліцензія
Авторське право (c) 2023 Assel Mukasheva, Dina Koishiyeva, Zhanna Suimenbayeva, Sabina Rakhmetulayeva, Aigerim Bolshibayeva, Gulnar Sadikova

Ця робота ліцензується відповідно до Creative Commons Attribution 4.0 International License.
Закріплення та умови передачі авторських прав (ідентифікація авторства) здійснюється у Ліцензійному договорі. Зокрема, автори залишають за собою право на авторство свого рукопису та передають журналу право першої публікації цієї роботи на умовах ліцензії Creative Commons CC BY. При цьому вони мають право укладати самостійно додаткові угоди, що стосуються неексклюзивного поширення роботи у тому вигляді, в якому вона була опублікована цим журналом, але за умови збереження посилання на першу публікацію статті в цьому журналі.
Ліцензійний договір – це документ, в якому автор гарантує, що володіє усіма авторськими правами на твір (рукопис, статтю, тощо).
Автори, підписуючи Ліцензійний договір з ПП «ТЕХНОЛОГІЧНИЙ ЦЕНТР», мають усі права на подальше використання свого твору за умови посилання на наше видання, в якому твір опублікований. Відповідно до умов Ліцензійного договору, Видавець ПП «ТЕХНОЛОГІЧНИЙ ЦЕНТР» не забирає ваші авторські права та отримує від авторів дозвіл на використання та розповсюдження публікації через світові наукові ресурси (власні електронні ресурси, наукометричні бази даних, репозитарії, бібліотеки тощо).
За відсутності підписаного Ліцензійного договору або за відсутністю вказаних в цьому договорі ідентифікаторів, що дають змогу ідентифікувати особу автора, редакція не має права працювати з рукописом.
Важливо пам’ятати, що існує і інший тип угоди між авторами та видавцями – коли авторські права передаються від авторів до видавця. В такому разі автори втрачають права власності на свій твір та не можуть його використовувати в будь-який спосіб.









