Розробка ефективних навчальних методів для виявлення та сегментації моделей при аналізі дефекту міжпересердної перегороди
DOI:
https://doi.org/10.15587/1729-4061.2024.312621Ключові слова:
глибоке навчання, SegFormer, ехокардіографія, сегментація зображення, YOLOv7, збільшення данихАнотація
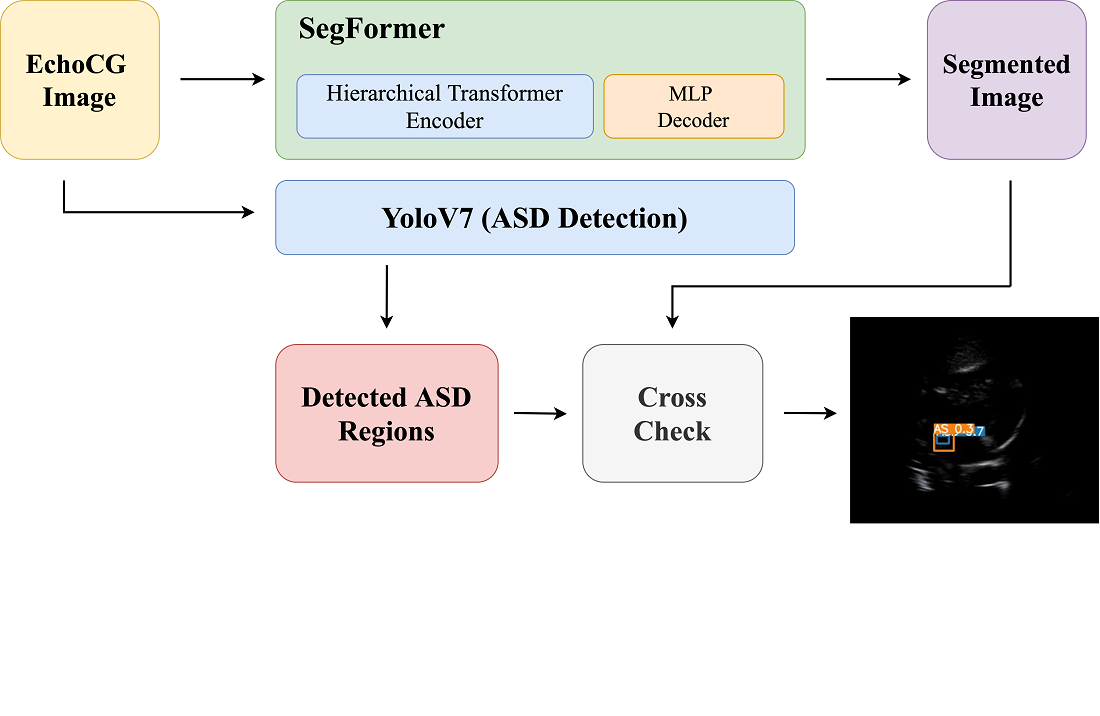
Метою цього дослідження є розробка ефективного конвеєра для виявлення дефектів міжпередсердної перегородки (ДМП) за допомогою ехокардіографічних зображень. ДМП — це поширені вроджені вади серця, які можуть призвести до серйозних проблем зі здоров’ям, якщо їх не діагностувати вчасно. Зростаючі показники смертності внаслідок невиявлених ДМП підкреслюють нагальну потребу у вдосконаленні методів діагностики. Щоб вирішити проблему обмежених анотованих медичних даних, які перешкоджають точним моделям виявлення, у цьому дослідженні було налаштовано модель SegFormer для точної сегментації серцевих структур на ехокардіографічних зображеннях, зосередившись на чотирикамерному вигляді серця, необхідному для виявлення ДМП. Завдяки інтеграції SegFormer з моделлю виявлення YOLOv7, відомою для виявлення об’єктів у реальному часі, області ДМП у сегментованих структурах серця були точно ідентифіковані. Це перехресне посилання забезпечує анатомічно точні діагнози та зменшує помилкові спрацьовування. Результати дослідження демонструють, що, незважаючи на обмежені дані, інтегрований метод досягає високої точності та швидкості, перевершуючи традиційні моделі. Це покращення пояснюється синергією між трансформаторною сегментацією SegFormer і ефективними можливостями виявлення YOLOv7. Відмінною рисою нашого підходу є успішна інтеграція цих моделей у спосіб, ефективний для даних, що дозволяє ефективно виявляти ДМП навіть за обмежених даних. Сфера практичного використання включає розгортання в клінічних умовах з обмеженими ресурсами, що вимагає лише ехокардіографічного обладнання та основних обчислювальних ресурсів. Надаючи клініцистам надійний інструмент для виявлення ДМП, дослідження підтримує своєчасне втручання в педіатричну кардіологію, в кінцевому підсумку покращуючи результати пацієнтів і підвищуючи послідовність лікування.
Посилання
- Williams, M. R., Perry, J. C. (2018). Arrhythmias and conduction disorders associated with atrial septal defects. Journal of Thoracic Disease, 10 (S24), S2940–S2944. https://doi.org/10.21037/jtd.2018.08.27
- Liu, Y., Huang, Q., Han, X., Liang, T., Zhang, Z., Lu, X. et al. (2024). Atrial Septal Defect Detection in Children Based on Ultrasound Video Using Multiple Instances Learning. Journal of Imaging Informatics in Medicine, 37 (3), 965–975. https://doi.org/10.1007/s10278-024-00987-1
- Mertens, L., Friedberg, M. K. (2009). The gold standard for noninvasive imaging in congenital heart disease: echocardiography. Current Opinion in Cardiology, 24 (2), 119–124. https://doi.org/10.1097/hco.0b013e328323d86f
- Sadeghpour, A., Alizadehasl, A. (2018). Echocardiography in the Critical Care Unit. Case-Based Textbook of Echocardiography, 423–430. https://doi.org/10.1007/978-3-319-67691-3_32
- Lancellotti, P., Price, S., Edvardsen, T., Cosyns, B., Neskovic, A. N., Dulgheru, R. et al. (2014). The use of echocardiography in acute cardiovascular care: Recommendations of the European Association of Cardiovascular Imaging and the Acute Cardiovascular Care Association. European Heart Journal - Cardiovascular Imaging, 16 (2), 119–146. https://doi.org/10.1093/ehjci/jeu210
- Tiver, K. D., Horsfall, M., Swan, A., De Pasquale, C., Horsfall, E., Chew, D. P., De Pasquale, C. G. (2022). Accuracy of Highly Limited Echocardiographic Screening Images for Determining a Structurally Normal Heart: The Quick-Six Study. Heart, Lung and Circulation, 31 (4), 462–468. https://doi.org/10.1016/j.hlc.2021.08.021
- Sicari, R., Gargani, L., Wiecek, A., Covic, A., Goldsmith, D., Suleymanlar, G. et al. (2012). The use of echocardiography in observational clinical trials: the EURECA-m registry. Nephrology Dialysis Transplantation, 28 (1), 19–23. https://doi.org/10.1093/ndt/gfs399
- Grenon, V., Szymonifka, J., Adler-Milstein, J., Ross, J., Sarkar, U. (2023). Factors Associated With Diagnostic Error: An Analysis of Closed Medical Malpractice Claims. Journal of Patient Safety, 19 (3), 211–215. https://doi.org/10.1097/pts.0000000000001105
- Li, Y., Liu, Z., Lai, Q., Li, S., Guo, Y., Wang, Y. et al. (2022). ESA-UNet for assisted diagnosis of cardiac magnetic resonance image based on the semantic segmentation of the heart. Frontiers in Cardiovascular Medicine, 9. https://doi.org/10.3389/fcvm.2022.1012450
- Madani, A., Arnaout, R., Mofrad, M., Arnaout, R. (2018). Fast and accurate view classification of echocardiograms using deep learning. Npj Digital Medicine, 1 (1). https://doi.org/10.1038/s41746-017-0013-1
- Shamir, O. (2018). Are resnets provably better than linear predictors. arXiv. https://doi.org/10.48550/arXiv.1804.06739
- Li, Y.-Z., Wang, Y., Huang, Y.-H., Xiang, P., Liu, W.-X., Lai, Q.-Q. et al. (2023). RSU-Net: U-net based on residual and self-attention mechanism in the segmentation of cardiac magnetic resonance images. Computer Methods and Programs in Biomedicine, 231, 107437. https://doi.org/10.1016/j.cmpb.2023.107437
- Ghorbani, A., Ouyang, D., Abid, A., He, B., Chen, J. H., Harrington, R. A. et al. (2020). Deep learning interpretation of echocardiograms. Npj Digital Medicine, 3 (1). https://doi.org/10.1038/s41746-019-0216-8
- Lim, G. B. (2020). Estimating ejection fraction by video-based AI. Nature Reviews Cardiology, 17 (6), 320–320. https://doi.org/10.1038/s41569-020-0375-y
- Guo, Z., Zhang, Y., Qiu, Z., Dong, S., He, S., Gao, H. et al. (2023). An improved contrastive learning network for semi-supervised multi-structure segmentation in echocardiography. Frontiers in Cardiovascular Medicine, 10. https://doi.org/10.3389/fcvm.2023.1266260
- Vakanski, A., Xian, M. (2021). Evaluation of Complexity Measures for Deep Learning Generalization in Medical Image Analysis. 2021 IEEE 31st International Workshop on Machine Learning for Signal Processing (MLSP), abs 2012 4115, 1–6. https://doi.org/10.1109/mlsp52302.2021.9596501
- Moradi, S., Oghli, M. G., Alizadehasl, A., Shiri, I., Oveisi, N., Oveisi, M. et al. (2019). MFP-Unet: A novel deep learning based approach for left ventricle segmentation in echocardiography. Physica Medica, 67, 58–69. https://doi.org/10.1016/j.ejmp.2019.10.001
- Xie, E., Wang, W., Yu, Z., Anandkumar, A., Alvarez, J. M., Luo, P. (2021). Segformer: Simple and efficient design for semantic segmentation with transformers. arXiv. https://doi.org/10.48550/arXiv.2105.15203
- Liu, X., Yang, X. (2008). Automatic acquisition of the four-chamber view for 3D echocardiography. IEICE Electronics Express, 5 (9), 316–320. https://doi.org/10.1587/elex.5.316
- Ukibassov, B. M., Rakhmetulayeva, S. B., Zhanabekov, Zh. O., Bolshibayeva, A. K., Yasar, A.-U.-H. (2024). Implementation of Anatomy Constrained Contrastive Learning for Heart Chamber Segmentation. Procedia Computer Science, 238, 536–543. https://doi.org/10.1016/j.procs.2024.06.057
- Zhou, Q., Sun, Z., Wang, L., Kang, B., Zhang, S., Wu, X. (2023). Mixture lightweight transformer for scene understanding. Computers and Electrical Engineering, 108, 108698. https://doi.org/10.1016/j.compeleceng.2023.108698
- Silvestry, F. E., Cohen, M. S., Armsby, L. B., Burkule, N. J., Fleishman, C. E., Hijazi, Z. M., Lang, R. M. et al. (2015). Guidelines for the Echocardiographic Assessment of Atrial Septal Defect and Patent Foramen Ovale: From the American Society of Echocardiography and Society for Cardiac Angiography and Interventions. Journal of the American Society of Echocardiography, 28 (8), 910–958. https://doi.org/10.1016/j.echo.2015.05.015
- Rakhmetulayeva, S. B., Bolshibayeva, A. K., Mukasheva, A. K., Ukibassov, B. M., Zhanabekov, Zh. O., Diaz, D. (2023). Machine learning methods and algorithms for predicting congenital heart pathologies. 2023 IEEE 17th International Conference on Application of Information and Communication Technologies (AICT). https://doi.org/10.1109/aict59525.2023.10313184
- Jwaid, W. M., Al-Husseini, Z. S. M., Sabry, A. H. (2021). Development of brain tumor segmentation of magnetic resonance imaging (MRI) using U-Net deep learning. Eastern-European Journal of Enterprise Technologies, 4 (9 (112)), 23–31. https://doi.org/10.15587/1729-4061.2021.238957
##submission.downloads##
Опубліковано
Як цитувати
Номер
Розділ
Ліцензія
Авторське право (c) 2024 Sabina Rakhmetulayeva, Baubek Ukibassov, Zhandos Zhanabekov, Aigerim Bolshibayeva

Ця робота ліцензується відповідно до Creative Commons Attribution 4.0 International License.
Закріплення та умови передачі авторських прав (ідентифікація авторства) здійснюється у Ліцензійному договорі. Зокрема, автори залишають за собою право на авторство свого рукопису та передають журналу право першої публікації цієї роботи на умовах ліцензії Creative Commons CC BY. При цьому вони мають право укладати самостійно додаткові угоди, що стосуються неексклюзивного поширення роботи у тому вигляді, в якому вона була опублікована цим журналом, але за умови збереження посилання на першу публікацію статті в цьому журналі.
Ліцензійний договір – це документ, в якому автор гарантує, що володіє усіма авторськими правами на твір (рукопис, статтю, тощо).
Автори, підписуючи Ліцензійний договір з ПП «ТЕХНОЛОГІЧНИЙ ЦЕНТР», мають усі права на подальше використання свого твору за умови посилання на наше видання, в якому твір опублікований. Відповідно до умов Ліцензійного договору, Видавець ПП «ТЕХНОЛОГІЧНИЙ ЦЕНТР» не забирає ваші авторські права та отримує від авторів дозвіл на використання та розповсюдження публікації через світові наукові ресурси (власні електронні ресурси, наукометричні бази даних, репозитарії, бібліотеки тощо).
За відсутності підписаного Ліцензійного договору або за відсутністю вказаних в цьому договорі ідентифікаторів, що дають змогу ідентифікувати особу автора, редакція не має права працювати з рукописом.
Важливо пам’ятати, що існує і інший тип угоди між авторами та видавцями – коли авторські права передаються від авторів до видавця. В такому разі автори втрачають права власності на свій твір та не можуть його використовувати в будь-який спосіб.









