Генерація, оцінка і порівняння (псевдо) випадкових послідовностей на основі ДНК
DOI:
https://doi.org/10.15587/1729-4061.2024.314808Ключові слова:
ДНК, випадкові послідовності, екстрактори випадковості, статистичне тестування, оцінка подібності, k-мери, MinHashАнотація
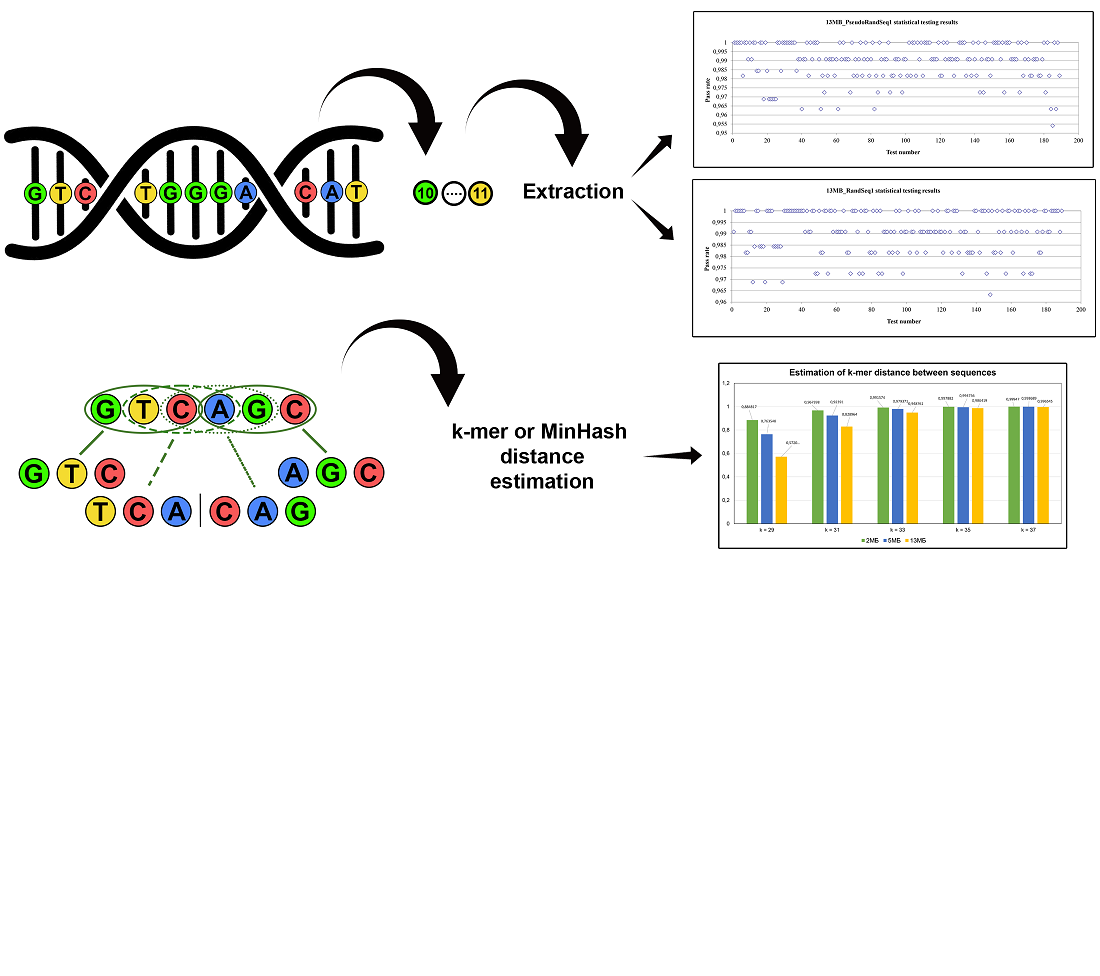
Об’єктом дослідження є методи та алгоритми обчислення, оцінки та порівняння псевдовипадкових (ПВП) і випадкових (ВП) послідовностей на основі ДНК. В роботі вирішується проблема екстракції (П)ВП з необхідними стохастичними та статистичними властивостями з ДНК-джерела шуму, експериментального підтвердження цих властивостей згідно вимог діючих міжнародних стандартів, а також оцінювання та порівняння отриманих для різних зразків ДНК послідовностей. Результатами є розроблені алгоритми генерації (П)ВП на основі ДНК, покращені алгоритми їх порівняння, а також пропозиції щодо алгоритму оцінки їх властивостей. Для вихідних даних реалізованих алгоритмів більше 96 % (як рекомендовано NIST) бітових потоків послідовностей успішно проходять статистичне тестування, а показники ентропії на біт ВП близькі до 1. Особливістю розроблених алгоритмів генерації є використання перевіреного компонента покращення – блокового шифру у режимі CTR – чим пояснюється можливість отримання унікальних випадкових даних з потрібними властивостями. Особливістю запропонованого алгоритму оцінювання є комплексність використовуваних тестів, а саме повна оцінка статистичних властивостей та показників ентропії. Особливостями покращених алгоритмів порівняння є забезпечення економії ресурсів та можливість оцінювання значно більших масивів даних. Це пояснюється застосуванням кращих за використанням пам’яті структур та типів даних і універсальних криптопримітивів із різними режимами.
Отримані результати можуть мати практичне застосування для генерації даних і оцінки їх відповідності для використання при обчисленні ключів та загальносистемних параметрів, виконанні перетворень у складі геш функцій, отриманні послідовностей довільного алфавіту, у протоколах з нульовим розголошенням, тощо
Посилання
- Goldreich, O. (2001). Foundations of Cryptography. Cambridge University Press. https://doi.org/10.1017/cbo9780511546891
- Matthias, P., Werner, S. (2023). A Proposal for Functionality Classes for Random Number Generators. Version 2.36 – Current intermediate document for the AIS 20/31 workshop. Available at: https://www.bsi.bund.de/SharedDocs/Downloads/EN/BSI/Certification/Interpretations/AIS_31_Functionality_classes_for_random_number_generators_e_2023.pdf?__blob=publicationFile&v=2
- Horbenko, I., Derevianko, Ya., Horbenko, D. (2024). DNK – dzherelo shumu ta nefizychnykh vypadkovykh poslidovnostei. Osnovni polozhennia praktychnykh doslidzhen. II Mizhnarodna naukovo-praktychna konferentsiya «Kiberborotba: rozvidka, zakhyst ta protydiya», 30–31. Available at: https://cyberwarfare.viti.edu.ua/assets/files/Cyberwarfare_2024.pdf
- Turan, M. S., Barker, E., Kelsey, J., McKay, K., Baish, M., Boyle, M. (2018). NIST SP 800-90B. Recommendation for the Entropy Sources Used for Random Bit Generation. NIST. https://doi.org/10.6028/NIST.SP.800-90B
- Rukhin, A., Soto, J., Nechvatal, J., Smid, M., Barker, E., Leigh, S. et al. (2010). NIST Special Publication 800-22 Revision 1a. A Statistical Test Suite for Random and Pseudorandom Number Generators for Cryptographic Applications. Available at: https://nvlpubs.nist.gov/nistpubs/Legacy/SP/nistspecialpublication800-22r1a.pdf
- Extractors and Pseudorandom Generators (2001). Journal of the ACM (JACM), 48 (4), 860–879. https://doi.org/10.1145/502090.502099
- Barman, P., Djilali, B., Benatmane, S., Nuh, A. (2024). A New Hybrid Cryptosystem Involving DNA, Rabin, One Time Pad and Fiestel. SSRN Electronic Journal. https://doi.org/10.2139/ssrn.4771411
- Zhang, Y., Liu, X., Sun, M. (2017). DNA based random key generation and management for OTP encryption. Biosystems, 159, 51–63. https://doi.org/10.1016/j.biosystems.2017.07.002
- Siddaramappa, V., Ramesh, K. B. (2020). True Random Number Generation Based on DNA molecule Genetic Information (DNA-TRNG). Cryptology ePrint Archive, Paper 2020/745. Available at: https://eprint.iacr.org/2020/745.pdf
- Li, Z., Peng, C., Tan, W., Li, L. (2020). A Novel Chaos-Based Image Encryption Scheme by Using Randomly DNA Encode and Plaintext Related Permutation. Applied Sciences, 10 (21), 7469. https://doi.org/10.3390/app10217469
- Sievers, F., Higgins, D. G. (2017). Clustal Omega for making accurate alignments of many protein sequences. Protein Science, 27 (1), 135–145. https://doi.org/10.1002/pro.3290
- Tu, S.-L., Staheli, J. P., McClay, C., McLeod, K., Rose, T. M., Upton, C. (2018). Base-By-Base Version 3: New Comparative Tools for Large Virus Genomes. Viruses, 10 (11), 637. https://doi.org/10.3390/v10110637
- Beretta, S. (2019). Algorithms for Strings and Sequences: Pairwise Alignment. Encyclopedia of Bioinformatics and Computational Biology, 22–29. https://doi.org/10.1016/b978-0-12-809633-8.20317-8
- Needleman, S. B., Wunsch, C. D. (1970). A general method applicable to the search for similarities in the amino acid sequence of two proteins. Journal of Molecular Biology, 48 (3), 443–453. https://doi.org/10.1016/0022-2836(70)90057-4
- Shanker, K. P. S., Austin, J., Sherly, E. (2010). An Algorithm for alignment-free sequence comparison using Logical Match. 2010 The 2nd International Conference on Computer and Automation Engineering (ICCAE), 536–538. https://doi.org/10.1109/iccae.2010.5452072
- Wilkinson, S. (2022). Fast K-Mer Counting and Clustering for Biological Sequence Analysis. Available at: https://cran.r-project.org/web/packages/kmer/kmer.pdf
- Ondov, B. D., Treangen, T. J., Melsted, P., Mallonee, A. B., Bergman, N. H., Koren, S., Phillippy, A. M. (2016). Mash: fast genome and metagenome distance estimation using MinHash. Genome Biology, 17 (1). https://doi.org/10.1186/s13059-016-0997-x
- Broder, A. Z. (1997). On the resemblance and containment of documents. Proceedings. Compression and Complexity of SEQUENCES 1997 (Cat. No.97TB100171). https://doi.org/10.1109/sequen.1997.666900
- Barker, E., Kelsey, J. (2015). NIST SP 800-90A Rev. 1. Recommendation for Random Number Generation Using Deterministic Random Bit Generators. NIST. http://dx.doi.org/10.6028/NIST.SP.800-90Ar1
- Holubnychiy, D. Yu., Kandiy, S. O., Yesina, M. V., Gorbenko, D. Yu. (2024). Methods and means for analysing, evaluating and comparing properties of random sequences and random numbers. Radiotekhnika, 1 (216), 30–45. https://doi.org/10.30837/rt.2024.1.216.02
- Müller, S. (2024). Linux /dev/random – A New Approach. Available at: https://www.chronox.de/lrng/releases/v53/lrng-v53.pdf
- Index of /genbank. Available at: https://ftp.ncbi.nlm.nih.gov/genbank/
- DNA Data Bank of Japan. Available at: https://www.ddbj.nig.ac.jp/index-e.html
- Contreras Rodríguez, L., Madarro-Capó , E. J., Legón-Pérez , C. M., Rojas, O., Sosa-Gómez, G. (2021). Selecting an Effective Entropy Estimator for Short Sequences of Bits and Bytes with Maximum Entropy. Entropy, 23 (5), 561. https://doi.org/10.3390/e23050561
- Skorski, M. (2016). Improved Estimation of Collision Entropy in High and Low-Entropy Regimes and Applications to Anomaly Detection. Cryptology ePrint Archive, Paper 2016/1035. Available at: https://eprint.iacr.org/2016/1035
- Quantis QRNG USB. Available at: https://www.idquantique.com/random-number-generation/products/quantis-random-number-generator/
- Aparatnyi HVCh «Hriada-3». AT «IIT». Available at: https://iit.com.ua/index.php?page=itemdetails&p=3>ype=1&type=1&id=96
- Chi-Square Goodness-of-Fit Test. NIST Engineering Statistics Handbook. Available at: https://www.itl.nist.gov/div898/handbook/eda/section3/eda35f.htm
- Genome Assembly Workshop 2020. UCDavis Bioinformatics Core. Available at: https://ucdavis-bioinformatics-training.github.io/2020-Genome_Assembly_Workshop/kmers/kmers
- Edgar, R. C. (2004). Local homology recognition and distance measures in linear time using compressed amino acid alphabets. Nucleic Acids Research, 32 (1), 380–385. https://doi.org/10.1093/nar/gkh180
- Kelsey, J. (2005). SHA-160: A Truncation Mode for SHA256 (and most other hashes). NIST. Available at: https://csrc.nist.gov/csrc/media/events/first-cryptographic-hash-workshop/documents/kelsey_truncation.pdf
- Dang, Q. (2009). NIST Special Publication 800-107. Recommendation for Applications Using Approved Hash Algorithms. NIST. Available at: https://csrc.nist.rip/library/NIST%20SP%20800-107%20Recommendation%20for%20Apps%20Using%20Approved%20Hash%20Algorithms,%202009-02.pdf
- Gorbenko, I. D., Alekseychuk, A. N., Kachko, Ye. G., Derevianko, Ya. A. (2024). Research into methods and algorithms for generating (pseudo) random sequences over an arbitrary alphabet. Radiotekhnika, 216, 7–29. https://doi.org/10.30837/rt.2024.1.216.01
##submission.downloads##
Опубліковано
Як цитувати
Номер
Розділ
Ліцензія
Авторське право (c) 2024 Ivan Gorbenko, Yaroslav Derevianko

Ця робота ліцензується відповідно до Creative Commons Attribution 4.0 International License.
Закріплення та умови передачі авторських прав (ідентифікація авторства) здійснюється у Ліцензійному договорі. Зокрема, автори залишають за собою право на авторство свого рукопису та передають журналу право першої публікації цієї роботи на умовах ліцензії Creative Commons CC BY. При цьому вони мають право укладати самостійно додаткові угоди, що стосуються неексклюзивного поширення роботи у тому вигляді, в якому вона була опублікована цим журналом, але за умови збереження посилання на першу публікацію статті в цьому журналі.
Ліцензійний договір – це документ, в якому автор гарантує, що володіє усіма авторськими правами на твір (рукопис, статтю, тощо).
Автори, підписуючи Ліцензійний договір з ПП «ТЕХНОЛОГІЧНИЙ ЦЕНТР», мають усі права на подальше використання свого твору за умови посилання на наше видання, в якому твір опублікований. Відповідно до умов Ліцензійного договору, Видавець ПП «ТЕХНОЛОГІЧНИЙ ЦЕНТР» не забирає ваші авторські права та отримує від авторів дозвіл на використання та розповсюдження публікації через світові наукові ресурси (власні електронні ресурси, наукометричні бази даних, репозитарії, бібліотеки тощо).
За відсутності підписаного Ліцензійного договору або за відсутністю вказаних в цьому договорі ідентифікаторів, що дають змогу ідентифікувати особу автора, редакція не має права працювати з рукописом.
Важливо пам’ятати, що існує і інший тип угоди між авторами та видавцями – коли авторські права передаються від авторів до видавця. В такому разі автори втрачають права власності на свій твір та не можуть його використовувати в будь-який спосіб.









