Удосконалення ехокардіографічної діагностики в реальному часі з гібридною моделлю глибокого навчання
DOI:
https://doi.org/10.15587/1729-4061.2024.314845Ключові слова:
глибоке навчання, машинне навчання, ЗНМ, YOLOv7, SegFormer, моделі на основі трансформераАнотація
Це дослідження зосереджено на розробці нової гібридної архітектури глибокого навчання, розробленої для аналізу ультразвукових зображень серця в реальному часі. Об’єктом дослідження є діагностична точність та ефективність виявлення таких патологій серця, як дефект міжпередсердної перегородки (ДМП) та стеноз аорти (СА) за даними УЗД.
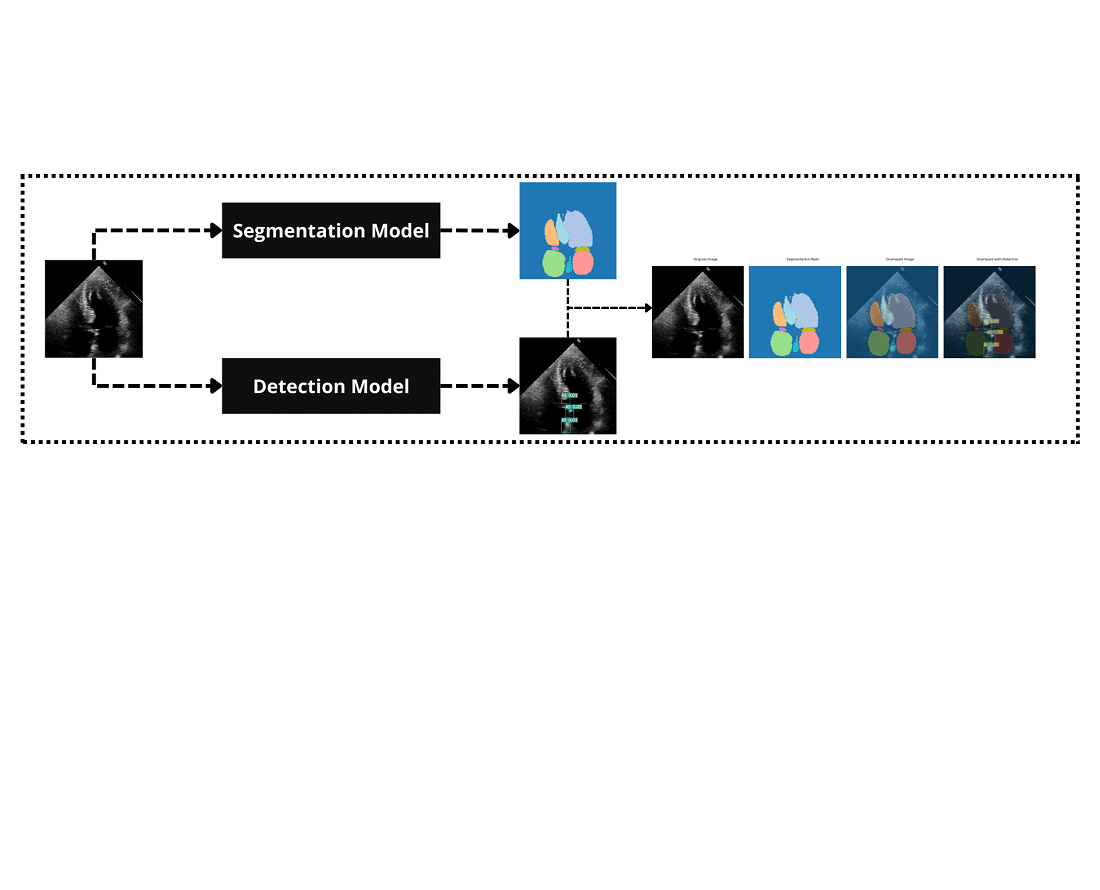
Проблема полягає в недостатній точності та можливості узагальнення існуючих моделей для аналізу зображень серця в реальному часі, що обмежує їх практичне клінічне застосування. Щоб вирішити цю проблему, згорткові нейронні мережі (ЗНМ), що поєднують виділення локальних ознак, були інтегровані з глобальним контекстним розумінням структур серця. Крім того, використовувався YOLOv7 для точної сегментації та виявлення.
Результати демонструють, що гібридна модель досягає загальної діагностичної точності 92 % для виявлення ДМП і 90 % для виявлення СА, що на 7 % перевищує стандартну модель YOLOv7. Ці вдосконалення пов’язані зі здатністю гібридної архітектури одночасно охоплювати дрібні анатомічні деталі та ширші структурні зв’язки, покращуючи виявлення тонких серцевих аномалій.
Отримані дані свідчать про те, що поєднання ЗНМ покращує розпізнавання образів і контекстний аналіз, що призводить до кращого виявлення серцевих аномалій. Ключові особливості, які сприяють вирішенню проблеми, включають здатність гібридної архітектури охоплювати детальні локальні особливості та ширший структурний контекст одночасно.
На практиці модель може бути застосована в клінічних умовах, де потрібна оцінка серця в режимі реального часу за допомогою стандартного медичного обладнання для візуалізації. Його обчислювальна ефективність і висока точність роблять його придатним навіть у середовищах з обмеженими ресурсами, скорочуючи час аналізу для клініцистів, підтримуючи персоналізовані плани лікування та потенційно покращуючи результати пацієнтів у кардіології
Посилання
- Cardiovascular diseases (CVDs) (2021). World Health Organization. Available at: https://www.who.int/news-room/fact-sheets/detail/cardiovascular-diseases-(cvds)
- Nagueh, S. F., Smiseth, O. A., Appleton, C. P., Byrd, B. F., Dokainish, H., Edvardsen, T. et al. (2016). Recommendations for the Evaluation of Left Ventricular Diastolic Function by Echocardiography: An Update from the American Society of Echocardiography and the European Association of Cardiovascular Imaging. European Heart Journal – Cardiovascular Imaging, 17 (12), 1321–1360. https://doi.org/10.1093/ehjci/jew082
- Zhou, J., Du, M., Chang, S., Chen, Z. (2021). Artificial intelligence in echocardiography: detection, functional evaluation, and disease diagnosis. Cardiovascular Ultrasound, 19 (1). https://doi.org/10.1186/s12947-021-00261-2
- Litjens, G., Kooi, T., Bejnordi, B. E., Setio, A. A. A., Ciompi, F., Ghafoorian, M. et al. (2017). A survey on deep learning in medical image analysis. Medical Image Analysis, 42, 60–88. https://doi.org/10.1016/j.media.2017.07.005
- Shen, D., Wu, G., Suk, H.-I. (2017). Deep Learning in Medical Image Analysis. Annual Review of Biomedical Engineering, 19 (1), 221–248. https://doi.org/10.1146/annurev-bioeng-071516-044442
- Wang, C.-Y., Bochkovskiy, A., Liao, H.-Y. M. (2023). YOLOv7: Trainable Bag-of-Freebies Sets New State-of-the-Art for Real-Time Object Detectors. 2023 IEEE/CVF Conference on Computer Vision and Pattern Recognition (CVPR), 7464–7475. https://doi.org/10.1109/cvpr52729.2023.00721
- Ho, J., Jain, A., Abbeel, P. (2020). Denoising diffusion probabilistic models. Advances in Neural Information Processing Systems, 33, 6840–6851.
- Zhang, J., Gajjala, S., Agrawal, P., Tison, G. H., Hallock, L. A., Beussink-Nelson, L. et al. (2018). Fully Automated Echocardiogram Interpretation in Clinical Practice. Circulation, 138 (16), 1623–1635. https://doi.org/10.1161/circulationaha.118.034338
- Ouyang, D., He, B., Ghorbani, A., Yuan, N., Ebinger, J., Langlotz, C. P. et al. (2020). Video-based AI for beat-to-beat assessment of cardiac function. Nature, 580 (7802), 252–256. https://doi.org/10.1038/s41586-020-2145-8
- Leclerc, S., Smistad, E., Pedrosa, J., Ostvik, A., Cervenansky, F., Espinosa, F. et al. (2019). Deep Learning for Segmentation Using an Open Large-Scale Dataset in 2D Echocardiography. IEEE Transactions on Medical Imaging, 38 (9), 2198–2210. https://doi.org/10.1109/tmi.2019.2900516
- Lang, R. M., Badano, L. P., Mor-Avi, V., Afilalo, J., Armstrong, A., Ernande, L. et al. (2015). Recommendations for Cardiac Chamber Quantification by Echocardiography in Adults: An Update from the American Society of Echocardiography and the European Association of Cardiovascular Imaging. Journal of the American Society of Echocardiography, 28 (1), 1-39.e14. https://doi.org/10.1016/j.echo.2014.10.003
- Knackstedt, C., Bekkers, S. C. A. M., Schummers, G., Schreckenberg, M., Muraru, D., Badano, L. P. et al. (2015). Fully Automated Versus Standard Tracking of Left Ventricular Ejection Fraction and Longitudinal Strain. Journal of the American College of Cardiology, 66 (13), 1456–1466. https://doi.org/10.1016/j.jacc.2015.07.052
- Madani, A., Arnaout, R., Mofrad, M., Arnaout, R. (2018). Fast and accurate view classification of echocardiograms using deep learning. Npj Digital Medicine, 1 (1). https://doi.org/10.1038/s41746-017-0013-1
- Ronneberger, O., Fischer, P., Brox, T. (2015). U-Net: Convolutional Networks for Biomedical Image Segmentation. Medical Image Computing and Computer-Assisted Intervention – MICCAI 2015, 234–241. https://doi.org/10.1007/978-3-319-24574-4_28
- Oktay, O., Schlemper, J., Folgoc, L. L., Lee, M., Heinrich, M., Misawa, K. et al. (2018). Attention U-Net: Learning where to look for the pancreas. arXiv. https://doi.org/10.48550/arXiv.1804.03999
- Wang, K., Wang, S., Xiong, M., Wang, C., Wang, H. (2021). Non-invasive Assessment of Hepatic Venous Pressure Gradient (HVPG) Based on MR Flow Imaging and Computational Fluid Dynamics. Medical Image Computing and Computer Assisted Intervention – MICCAI 2021, 33–42. https://doi.org/10.1007/978-3-030-87234-2_4
- Mudeng, V., Nisa, W., Sukmananda Suprapto, S. (2022). Computational image reconstruction for multi-frequency diffuse optical tomography. Journal of King Saud University - Computer and Information Sciences, 34 (6), 3527–3538. https://doi.org/10.1016/j.jksuci.2020.12.015
- Nurmaini, S., Sapitri, A. I., Tutuko, B., Rachmatullah, M. N., Rini, D. P., Darmawahyuni, A. et al. (2023). Automatic echocardiographic anomalies interpretation using a stacked residual-dense network model. BMC Bioinformatics, 24 (1). https://doi.org/10.1186/s12859-023-05493-9
- Iriani Sapitri, A., Nurmaini, S., Naufal Rachmatullah, M., Tutuko, B., Darmawahyuni, A., Firdaus, F. et al. (2023). Deep learning-based real time detection for cardiac objects with fetal ultrasound video. Informatics in Medicine Unlocked, 36, 101150. https://doi.org/10.1016/j.imu.2022.101150
- Sutarno, S., Nurmaini, S., Partan, R. U., Sapitri, A. I., Tutuko, B., Naufal Rachmatullah, M. et al. (2022). FetalNet: Low-light fetal echocardiography enhancement and dense convolutional network classifier for improving heart defect prediction. Informatics in Medicine Unlocked, 35, 101136. https://doi.org/10.1016/j.imu.2022.101136
- Saxena, A., Singh, S. P., Gaidhane, V. H. (2022). A deep learning approach for the detection of COVID-19 from chest X-ray images using convolutional neural networks. Advances in Machine Learning & Artificial Intelligence, 3 (2), 52–65. https://doi.org/10.33140/amlai.03.02.01
- Zhou, Q., Sun, Z., Wang, L., Kang, B., Zhang, S., Wu, X. (2023). Mixture lightweight transformer for scene understanding. Computers and Electrical Engineering, 108, 108698. https://doi.org/10.1016/j.compeleceng.2023.108698
- Xie, E., Wang, W., Yu, Z., Anandkumar, A., Alvarez, J. M., Luo, P. (2021). SegFormer: Simple and efficient design for semantic segmentation with transformers. arXiv. https://doi.org/10.48550/arXiv.2105.15203
- Rakhmetulayeva, S. B., Bolshibayeva, A. K., Mukasheva, A. K., Ukibassov, B. M., Zhanabekov, Zh. O., Diaz, D. (2023). Machine learning methods and algorithms for predicting congenital heart pathologies. 2023 IEEE 17th International Conference on Application of Information and Communication Technologies (AICT). https://doi.org/10.1109/aict59525.2023.10313184
- Ukibassov, B. M., Rakhmetulayeva, S. B., Zhanabekov, Zh. O., Bolshibayeva, A. K., Yasar, A.-U.-H. (2024). Implementation of Anatomy Constrained Contrastive Learning for Heart Chamber Segmentation. Procedia Computer Science, 238, 536–543. https://doi.org/10.1016/j.procs.2024.06.057
- Rakhmetulayeva, S., Syrymbet, Z. (2022). Implementation of convolutional neural network for predicting glaucoma from fundus images. Eastern-European Journal of Enterprise Technologies, 6 (2 (120)), 70–77. https://doi.org/10.15587/1729-4061.2022.269229
- He, K., Zhang, X., Ren, S., Sun, J. (2016). Deep Residual Learning for Image Recognition. 2016 IEEE Conference on Computer Vision and Pattern Recognition (CVPR). https://doi.org/10.1109/cvpr.2016.90
- Liu, Z., Lin, Y., Cao, Y., Hu, H., Wei, Y., Zhang, Z. et al. (2021). Swin Transformer: Hierarchical Vision Transformer using Shifted Windows. 2021 IEEE/CVF International Conference on Computer Vision (ICCV), 9992–10002. https://doi.org/10.1109/iccv48922.2021.00986
- Ren, S., He, K., Girshick, R., Sun, J. (2017). Faster R-CNN: Towards Real-Time Object Detection with Region Proposal Networks. IEEE Transactions on Pattern Analysis and Machine Intelligence, 39 (6), 1137–1149. https://doi.org/10.1109/tpami.2016.2577031
- LLiu, W., Anguelov, D., Erhan, D., Szegedy, C., Reed, S., Fu, C.-Y., Berg, A. C. (2016). SSD: Single Shot MultiBox Detector. Computer Vision – ECCV 2016, 21–37. https://doi.org/10.1007/978-3-319-46448-0_2
- Ba, J. L., Kiros, J. R., Hinton, G. E. (2016). Layer normalization. arXiv. https://doi.org/10.48550/arXiv.1607.06450
- Loshchilov, I., Hutter, F. (2019). Decoupled weight decay regularization. arXiv. https://doi.org/10.48550/arXiv.1711.05101
##submission.downloads##
Опубліковано
Як цитувати
Номер
Розділ
Ліцензія
Авторське право (c) 2024 Aigerim Bolshibayeva, Sabina Rakhmetulayeva, Baubek Ukibassov, Zhandos Zhanabekov

Ця робота ліцензується відповідно до Creative Commons Attribution 4.0 International License.
Закріплення та умови передачі авторських прав (ідентифікація авторства) здійснюється у Ліцензійному договорі. Зокрема, автори залишають за собою право на авторство свого рукопису та передають журналу право першої публікації цієї роботи на умовах ліцензії Creative Commons CC BY. При цьому вони мають право укладати самостійно додаткові угоди, що стосуються неексклюзивного поширення роботи у тому вигляді, в якому вона була опублікована цим журналом, але за умови збереження посилання на першу публікацію статті в цьому журналі.
Ліцензійний договір – це документ, в якому автор гарантує, що володіє усіма авторськими правами на твір (рукопис, статтю, тощо).
Автори, підписуючи Ліцензійний договір з ПП «ТЕХНОЛОГІЧНИЙ ЦЕНТР», мають усі права на подальше використання свого твору за умови посилання на наше видання, в якому твір опублікований. Відповідно до умов Ліцензійного договору, Видавець ПП «ТЕХНОЛОГІЧНИЙ ЦЕНТР» не забирає ваші авторські права та отримує від авторів дозвіл на використання та розповсюдження публікації через світові наукові ресурси (власні електронні ресурси, наукометричні бази даних, репозитарії, бібліотеки тощо).
За відсутності підписаного Ліцензійного договору або за відсутністю вказаних в цьому договорі ідентифікаторів, що дають змогу ідентифікувати особу автора, редакція не має права працювати з рукописом.
Важливо пам’ятати, що існує і інший тип угоди між авторами та видавцями – коли авторські права передаються від авторів до видавця. В такому разі автори втрачають права власності на свій твір та не можуть його використовувати в будь-який спосіб.









