Гібридна імпутація біомедичних даних на основі трансформерів та автоенкодерів для оцінки біологічного віку людини
DOI:
https://doi.org/10.15587/1729-4061.2025.340325Ключові слова:
відновлення пропусків, гібридна архітектура, глибинне навчання, функціональний вік, PhenoAge, NHANESАнотація
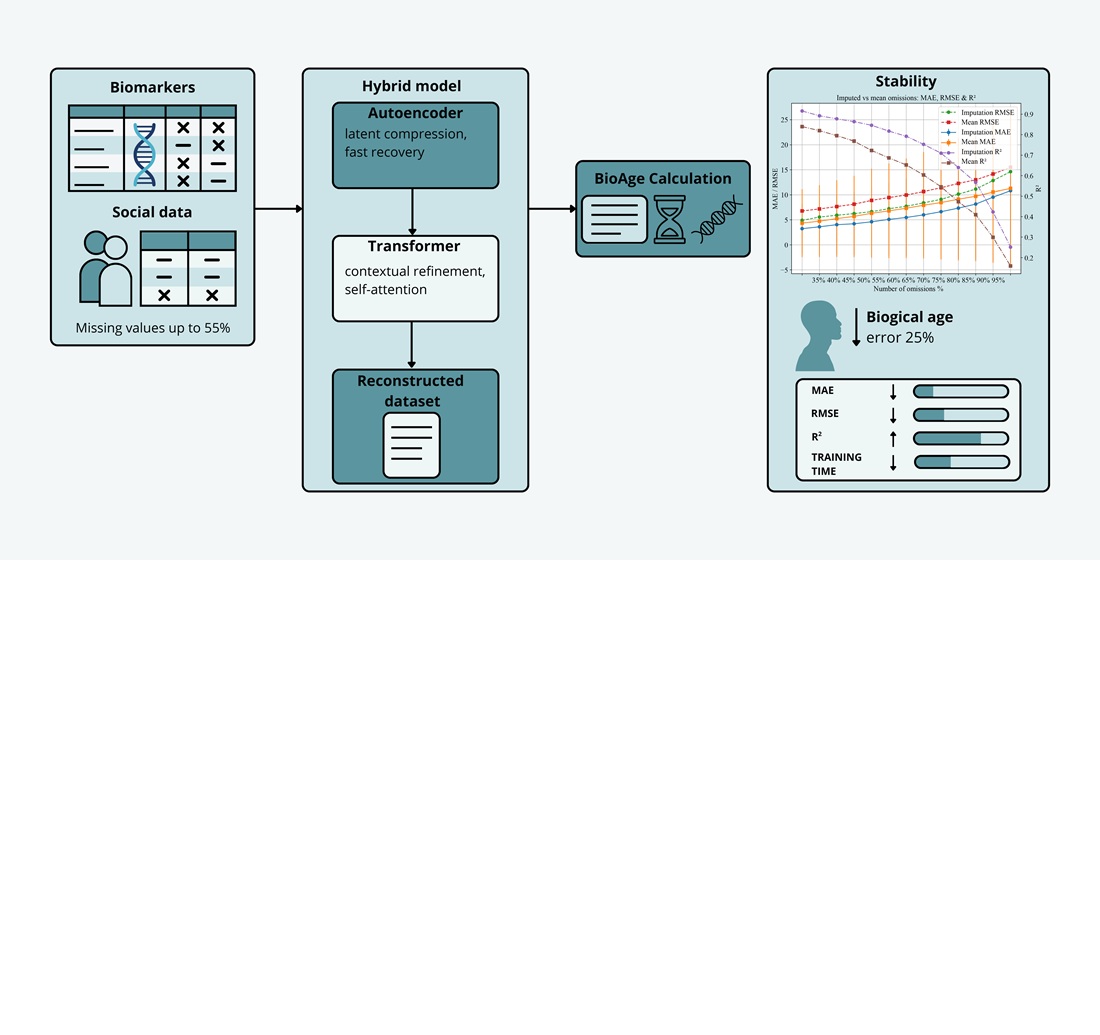
Об’єктом дослідження є процес відновлення пропущених біомедичних і соціальних даних для оцінки біологічного віку людини. Проблема полягає у великій кількості пропусків у наборах, зокрема NHANES – до 40%. Це знижує точність прогнозів здоров’я та ефективність профілактики. В дослідженні було використано методи глибинного навчання – автоенкодери та трансформери. Автоенкодер забезпечив швидку імпутацію (37.4 с, MAE = 7.54), але поступився точністю. Трансформер досяг найменшої похибки (246.3 с, MAE = 1.10), однак вимагав значних ресурсів і мав ризик перенавчання. Запропоновано гібридну архітектуру, що поєднує переваги підходів. На наборі NHANES (55081 спостережень, 84 біомаркери) вона показала оптимальний баланс (54.2 с, MAE = 5.26). Модель стабільна при 50% пропущених даних і підвищує точність оцінки біологічного віку на 25% порівняно із заповненням середнім значенням. Переваги пояснюються поєднанням швидкодії автоенкодера та контекстної чутливості трансформера. Показано, що для гібридної моделі R2 = 0.9875, а RMSE = 35.9, що свідчить про високу узгодженість відновлених значень. Аналіз чутливості підтвердив збереження точності при збільшенні частки пропусків до критичного рівня 55%, після якого відбувається деградація моделі. Унікальною рисою гібридного підходу є поєднання високої точності та помірних обчислювальних витрат. Це робить модель придатною для медичних інформаційних систем із неповними даними. Практичне застосування – профілактична медицина, оцінка старіння та виявлення груп ризику. В Україні модель може підвищити ефективність медичних досліджень та цифрової медицини. Вона також здатна стати основою для майбутніх досліджень у сфері біоінформатики та прогнозування тривалості життя
Посилання
- Poliahushko, L., Volkov, O. (2024). Socioeconomic influence on biological age: an overview of current studies and role of artificial intelligence. Telecommunication and information technologies, 3 (84), 120–130. https://doi.org/10.31673/2412-4338.2024.03041234
- Lau, D. T., Ahluwalia, N., Fryar, C. D., Kaufman, M., Arispe, I. E., Paulose-Ram, R. (2023). Data Related to Social Determinants of Health Captured in the National Health and Nutrition Examination Survey. American Journal of Public Health, 113 (12), 1290–1295. https://doi.org/10.2105/ajph.2023.307490
- Kowsar, I., Rabbani, S. B., Samad, M. D. (2024). Attention-Based Imputation of Missing Values in Electronic Health Records Tabular Data. 2024 IEEE 12th International Conference on Healthcare Informatics (ICHI), 177–182. https://doi.org/10.1109/ichi61247.2024.00030
- Casella, M., Milano, N., Dolce, P., Marocco, D. (2024). Transformers deep learning models for missing data imputation: an application of the ReMasker model on a psychometric scale. Frontiers in Psychology, 15. https://doi.org/10.3389/fpsyg.2024.1449272
- Lim, D. K., Rashid, N. U., Oliva, J. B., Ibrahim, J. G. (2024). Unsupervised Imputation of Non-Ignorably Missing Data Using Importance-Weighted Autoencoders. Statistics in Biopharmaceutical Research, 17 (2), 222–234. https://doi.org/10.1080/19466315.2024.2368787
- Horvath, S. (2013). DNA methylation age of human tissues and cell types. Genome Biology, 14 (10). https://doi.org/10.1186/gb-2013-14-10-r115
- Levine, M. E., Lu, A. T., Quach, A., Chen, B. H., Assimes, T. L., Bandinelli, S. et al. (2018). An epigenetic biomarker of aging for lifespan and healthspan. Aging, 10 (4), 573–591. https://doi.org/10.18632/aging.101414
- Aracri, F., Bianco, M. G., Quattrone, A., Sarica, A. (2025). Bridging the Gap: Missing Data Imputation Methods and Their Effect on Dementia Classification Performance. Brain Sciences, 15 (6), 639. https://doi.org/10.3390/brainsci15060639
- Altamimi, A., Alarfaj, A. A., Umer, M., Alabdulqader, E. A., Alsubai, S., Kim, T., Ashraf, I. (2024). An automated approach to predict diabetic patients using KNN imputation and effective data mining techniques. BMC Medical Research Methodology, 24 (1). https://doi.org/10.1186/s12874-024-02324-0
- Madley-Dowd, P., Curnow, E., Hughes, R. A., Cornish, R. P., Tilling, K., Heron, J. (2024). Analyses using multiple imputation need to consider missing data in auxiliary variables. American Journal of Epidemiology, 194 (6), 1756–1763. https://doi.org/10.1093/aje/kwae306
- Beaulieu-Jones, B. K., Moore, J. H. (2017). Missing data imputation in the electronic health record using deeply learned autoencoders. Biocomputing 2017, 207–218. https://doi.org/10.1142/9789813207813_0021
- Gondara, L., Wang, K. (2018). MIDA: Multiple Imputation Using Denoising Autoencoders. Advances in Knowledge Discovery and Data Mining, 260–272. https://doi.org/10.1007/978-3-319-93040-4_21
- Li, Y., Rao, S., Solares, J. R. A., Hassaine, A., Ramakrishnan, R., Canoy, D. et al. (2020). BEHRT: Transformer for Electronic Health Records. Scientific Reports, 10 (1). https://doi.org/10.1038/s41598-020-62922-y
- Khan, M. A. (2024). A Comparative Study on Imputation Techniques: Introducing a Transformer Model for Robust and Efficient Handling of Missing EEG Amplitude Data. Bioengineering, 11 (8), 740. https://doi.org/10.3390/bioengineering11080740
- He, S., Grant, P. E., Ou, Y. (2022). Global-Local Transformer for Brain Age Estimation. IEEE Transactions on Medical Imaging, 41 (1), 213–224. https://doi.org/10.1109/tmi.2021.3108910
- Urban, A., Sidorenko, D., Zagirova, D., Kozlova, E., Kalashnikov, A., Pushkov, S. et al. (2023). Precious1GPT: multimodal transformer-based transfer learning for aging clock development and feature importance analysis for aging and age-related disease target discovery. Aging. https://doi.org/10.18632/aging.204788
- Wang, X., Chen, H., Zhang, J., Fan, J. (2024). Generative adversarial learning for missing data imputation. Neural Computing and Applications, 37 (3), 1403–1416. https://doi.org/10.1007/s00521-024-10652-x
- Hong, S., Lynn, H. S. (2020). Accuracy of random-forest-based imputation of missing data in the presence of non-normality, non-linearity, and interaction. BMC Medical Research Methodology, 20 (1). https://doi.org/10.1186/s12874-020-01080-1
- Zhou, Y.-H., Saghapour, E. (2021). ImputEHR: A Visualization Tool of Imputation for the Prediction of Biomedical Data. Frontiers in Genetics, 12. https://doi.org/10.3389/fgene.2021.691274
- Bae, C.-Y., Im, Y., Lee, J., Park, C.-S., Kim, M., Kwon, H. et al. (2021). Comparison of Biological Age Prediction Models Using Clinical Biomarkers Commonly Measured in Clinical Practice Settings: AI Techniques Vs. Traditional Statistical Methods. Frontiers in Analytical Science, 1. https://doi.org/10.3389/frans.2021.709589
- United States Department of Health and Human Services. Centers for Disease Control and Prevention. National Center for Health Statistics. National Health and Nutrition Examination Survey (NHANES), 1999-2000 (2012). Inter-university Consortium for Political and Social Research [distributor]. https://doi.org/10.3886/icpsr25501.v4
- Mack, C., Su, Z., Weistreich, D. (2018). Managing Missing Data in Patient Registries. Agency for Healthcare Research and Quality (AHRQ). https://doi.org/10.23970/ahrqregistriesmissingdata
- Chicco, D., Warrens, M. J., Jurman, G. (2021). The coefficient of determination R-squared is more informative than SMAPE, MAE, MAPE, MSE and RMSE in regression analysis evaluation. PeerJ Computer Science, 7, e623. https://doi.org/10.7717/peerj-cs.623
- da Silva, I. N., Hernane Spatti, D., Andrade Flauzino, R., Liboni, L. H. B., dos Reis Alves, S. F. (2016). Multilayer Perceptron Networks. Artificial Neural Networks, 55–115. https://doi.org/10.1007/978-3-319-43162-8_5
- Jinbo, Z., Yufu, L., Haitao, M. (2025). Handling missing data of using the XGBoost-based multiple imputation by chained equations regression method. Frontiers in Artificial Intelligence, 8. https://doi.org/10.3389/frai.2025.1553220
##submission.downloads##
Опубліковано
Як цитувати
Номер
Розділ
Ліцензія
Авторське право (c) 2025 Volodymyr Slipchenko, Liubov Poliahushko, Oleksandr Volkov, Vladyslav Shatylo

Ця робота ліцензується відповідно до Creative Commons Attribution 4.0 International License.
Закріплення та умови передачі авторських прав (ідентифікація авторства) здійснюється у Ліцензійному договорі. Зокрема, автори залишають за собою право на авторство свого рукопису та передають журналу право першої публікації цієї роботи на умовах ліцензії Creative Commons CC BY. При цьому вони мають право укладати самостійно додаткові угоди, що стосуються неексклюзивного поширення роботи у тому вигляді, в якому вона була опублікована цим журналом, але за умови збереження посилання на першу публікацію статті в цьому журналі.
Ліцензійний договір – це документ, в якому автор гарантує, що володіє усіма авторськими правами на твір (рукопис, статтю, тощо).
Автори, підписуючи Ліцензійний договір з ПП «ТЕХНОЛОГІЧНИЙ ЦЕНТР», мають усі права на подальше використання свого твору за умови посилання на наше видання, в якому твір опублікований. Відповідно до умов Ліцензійного договору, Видавець ПП «ТЕХНОЛОГІЧНИЙ ЦЕНТР» не забирає ваші авторські права та отримує від авторів дозвіл на використання та розповсюдження публікації через світові наукові ресурси (власні електронні ресурси, наукометричні бази даних, репозитарії, бібліотеки тощо).
За відсутності підписаного Ліцензійного договору або за відсутністю вказаних в цьому договорі ідентифікаторів, що дають змогу ідентифікувати особу автора, редакція не має права працювати з рукописом.
Важливо пам’ятати, що існує і інший тип угоди між авторами та видавцями – коли авторські права передаються від авторів до видавця. В такому разі автори втрачають права власності на свій твір та не можуть його використовувати в будь-який спосіб.









