Розробка комплексного підходу до автоматичного діагностування підтипів раку молочної залози на основі глибоких нейронних мереж
DOI:
https://doi.org/10.15587/1729-4061.2025.344041Ключові слова:
Attention U-Net, генетичний алгоритм, оптимізація архітектури нейронної мережі, ІГХ-зображення, сегментація біомедичних зображень, автоматичне діагностування раку молочної залозиАнотація
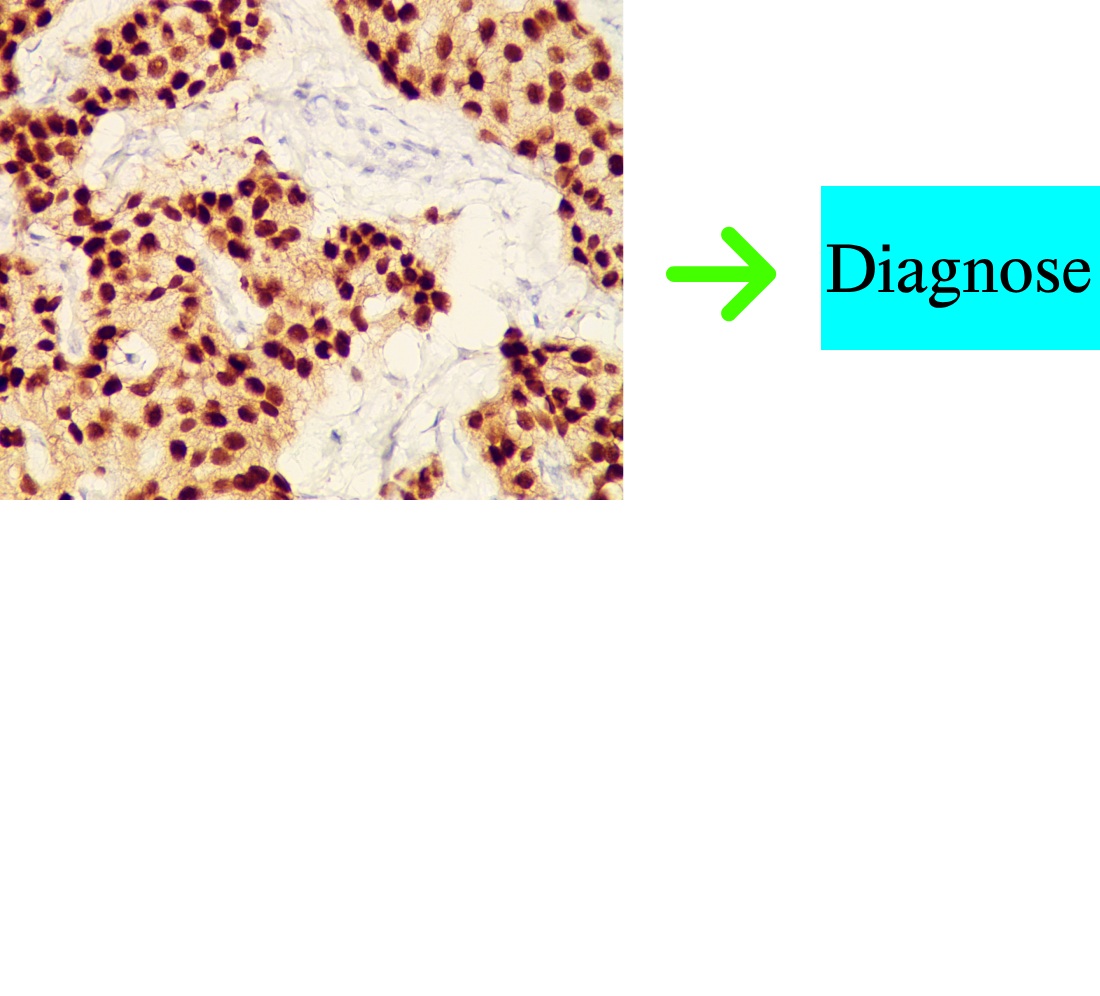
Об’єктом дослідження є процес аналізу імуногістохімічних зображень раку молочної залози. Дослідження дозволило внести свій вклад у вирішення проблеми стандартизованого та об’єктивного підходу до кількісної оцінки імуногістохімічних біомаркерів, що мінімізував би міжособистісну варіабельність оцінок та був обчислювально ефективним для аналізу біомедичних зображень.
Дослідження присвячене пошуку балансу між складністю моделі та узагальненням з використанням еволюційних алгоритмів для налаштування глибоких нейронних мереж для біомедичних завдань, аналізуючи вплив структури мережі на продуктивність.
У роботі було проведено експерименти із сегментації імуногістохімічних зображень на 13 різних архітектурах нейронних мереж. Оцінювання здійснювалося за допомогою п’яти метрик точності, що дозволило об’єктивно порівняти продуктивність моделей. Використання генетичного алгоритму для оптимізації архітектури нейронної мережі дозволило адаптивно знаходити комбінації параметрів, зокрема кількість шарів та розмір базового фільтра. Еволюційний підхід забезпечив ефективне дослідження простору конфігурацій, що призвело до підвищення метрики Dice до 0.74. Отримане зростання точності свідчить про покращену здатність моделі до сегментації зображень із різними характеристиками, що демонструє практичну ефективність запропонованого підходу для задач біомедичного діагностування.
Оптимізовану архітектуру застосовано для розробки системи автоматичного діагностування раку молочної залози на основі нейронних мереж, зокрема для методу автоматичного діагностування підтипів раку молочної залози. Це посприяло підвищенню точності аналізу біомедичних зображень, що допоможе покращити діагностичний процес у клінічній практиці
Посилання
- Hamet, P., Tremblay, J. (2017). Artificial intelligence in medicine. Metabolism, 69, S36–S40. https://doi.org/10.1016/j.metabol.2017.01.011
- Briganti, G., Le Moine, O. (2020). Artificial Intelligence in Medicine: Today and Tomorrow. Frontiers in Medicine, 7. https://doi.org/10.3389/fmed.2020.00027
- Litjens, G., Kooi, T., Bejnordi, B. E., Setio, A. A. A., Ciompi, F., Ghafoorian, M. et al. (2017). A survey on deep learning in medical image analysis. Medical Image Analysis, 42, 60–88. https://doi.org/10.1016/j.media.2017.07.005
- Srikantamurthy, M. M., Rallabandi, V. P. S., Dudekula, D. B., Natarajan, S., Park, J. (2023). Classification of benign and malignant subtypes of breast cancer histopathology imaging using hybrid CNN-LSTM based transfer learning. BMC Medical Imaging, 23 (1). https://doi.org/10.1186/s12880-023-00964-0
- Al-Jabbar, M., Alshahrani, M., Senan, E. M., Ahmed, I. A. (2023). Analyzing Histological Images Using Hybrid Techniques for Early Detection of Multi-Class Breast Cancer Based on Fusion Features of CNN and Handcrafted. Diagnostics, 13 (10), 1753. https://doi.org/10.3390/diagnostics13101753
- Miranda Ruiz, F., Lahrmann, B., Bartels, L., Krauthoff, A., Keil, A., Härtel, S. et al. (2023). CNN stability training improves robustness to scanner and IHC-based image variability for epithelium segmentation in cervical histology. Frontiers in Medicine, 10. https://doi.org/10.3389/fmed.2023.1173616
- Zaha, D. C. (2014). Significance of immunohistochemistry in breast cancer. World Journal of Clinical Oncology, 5 (3), 382. https://doi.org/10.5306/wjco.v5.i3.382
- Aswathy M. A., Mohan, J. (2020). Analysis of Machine Learning Algorithms for Breast Cancer Detection. Handbook of Research on Applications and Implementations of Machine Learning Techniques, 1–20. https://doi.org/10.4018/978-1-5225-9902-9.ch001
- Nabok, A. I. (2023). Prevalence and incidence of breast cancer in Ukraine. Wiadomości Lekarskie, 76 (10), 2219–2223. Available at: https://www.researchgate.net/profile/Serhii-Tertyshnyi/publication/375025887_WL_Layout_10_2023/links/653bdaf73cc79d48c5b14c25/WL-Layout-10-2023.pdf#page=93
- Siegel, R. L., Kratzer, T. B., Giaquinto, A. N., Sung, H., Jemal, A. (2025). Cancer statistics, 2025. CA: A Cancer Journal for Clinicians, 75 (1), 10–45. https://doi.org/10.3322/caac.21871
- Ronneberger, O., Fischer, P., Brox, T. (2015). U-Net: Convolutional Networks for Biomedical Image Segmentation. Medical Image Computing and Computer-Assisted Intervention – MICCAI 2015, 234–241. https://doi.org/10.1007/978-3-319-24574-4_28
- Polley, M.-Y. C., Leung, S. C. Y., McShane, L. M., Gao, D., Hugh, J. C., Mastropasqua, M. G. et al. (2013). An International Ki67 Reproducibility Study. JNCI: Journal of the National Cancer Institute, 105 (24), 1897–1906. https://doi.org/10.1093/jnci/djt306
- Kumar, N., Gupta, R., Gupta, S. (2020). Whole Slide Imaging (WSI) in Pathology: Current Perspectives and Future Directions. Journal of Digital Imaging, 33 (4), 1034–1040. https://doi.org/10.1007/s10278-020-00351-z
- Siddique, N., Paheding, S., Elkin, C. P., Devabhaktuni, V. (2021). U-Net and Its Variants for Medical Image Segmentation: A Review of Theory and Applications. IEEE Access, 9, 82031–82057. https://doi.org/10.1109/access.2021.3086020
- Mehta, R., Arbel, T. (2019). 3D U-Net for Brain Tumour Segmentation. Brainlesion: Glioma, Multiple Sclerosis, Stroke and Traumatic Brain Injuries, 254–266. https://doi.org/10.1007/978-3-030-11726-9_23
- Chen, W., Liu, B., Peng, S., Sun, J., Qiao, X. (2019). S3D-UNet: Separable 3D U-Net for Brain Tumor Segmentation. Brainlesion: Glioma, Multiple Sclerosis, Stroke and Traumatic Brain Injuries, 358–368. https://doi.org/10.1007/978-3-030-11726-9_32
- Benny, S., Varma, S. L. (2021). Semantic Segmentation in Immunohistochemistry Breast Cancer Image using Deep Learning. 2021 International Conference on Advances in Computing, Communication, and Control (ICAC3), 1–3. https://doi.org/10.1109/icac353642.2021.9697264
- Benny, S., Varma, S. L. (2023). Attention-enhanced residual U-Net for nucleus segmentation in immunohistochemistry images. International Journal of Applied Engineering & Technology, 5 (4), 1266–1283. Available at: https://romanpub.com/resources/ijaet20v5-4-2023-138.pdf
- Mahanta, L. B., Hussain, E., Das, N., Kakoti, L., Chowdhury, M. (2021). IHC-Net: A fully convolutional neural network for automated nuclear segmentation and ensemble classification for Allred scoring in breast pathology. Applied Soft Computing, 103, 107136. https://doi.org/10.1016/j.asoc.2021.107136
- Kromp, F., Fischer, L., Bozsaky, E., Ambros, I. M., Dorr, W., Beiske, K. et al. (2021). Evaluation of Deep Learning Architectures for Complex Immunofluorescence Nuclear Image Segmentation. IEEE Transactions on Medical Imaging, 40 (7), 1934–1949. https://doi.org/10.1109/tmi.2021.3069558
- Xu, S., Li, G., Song, H., Wang, J., Wang, Y., Li, Q. (2024). GeNSeg-Net: A General Segmentation Framework for Any Nucleus in Immunohistochemistry Images. Proceedings of the 32nd ACM International Conference on Multimedia, 4475–4484. https://doi.org/10.1145/3664647.3681441
- Aboudessouki, A., Ali, Kh. M., Elsharkawy, M., Alksas, A., Mahmoud, A., Khalifa, F. et al. (2023). Automated Diagnosis of Breast Cancer Using Deep Learning-Based Whole Slide Image Analysis of Molecular Biomarkers. 2023 IEEE International Conference on Image Processing (ICIP). https://doi.org/10.1109/icip49359.2023.10222479
- Tkachova, O. V., Melnyk, H. M., Pitsun, O. Y., Datsko, T. V., Klishch, I. M., Derysh, B. B. (2023). A. s. No. 118979. Baza danykh tsyfrovykh imunohistokhimichnykh zobrazhen raku molochnoi zalozy «IHCDBI». declareted: 10.05.2023; published: 31.07.2023, Bul. No. 76.
- Huynh, N. (2023). Understanding evaluation metrics in Medical Image Segmentation. Available at: https://medium.com/mastering-data-science/understanding-evaluation-metrics-in-medical-image-segmentation-d289a373a3f
- WBRT pislia BCS. Rak molochnoi zalozy na rannikh stadiyakh: Klinichna nastanova, zasnovana na dokazakh (2024). Ministerstvo okhorony zdorovia Ukrainy, 27–28. Available at: https://www.dec.gov.ua/wp-content/uploads/2025/02/kn_2025_rannij-rmz.pdf
- Berezsky, O., Pitsun, O., Melnyk, G., Datsko, T., Izonin, I., Derysh, B. (2023). An Approach toward Automatic Specifics Diagnosis of Breast Cancer Based on an Immunohistochemical Image. Journal of Imaging, 9 (1), 12. https://doi.org/10.3390/jimaging9010012
- Cardoso, F., Kyriakides, S., Ohno, S., Penault-Llorca, F., Poortmans, P., Rubio, I. T. et al. (2019). Early breast cancer: ESMO Clinical Practice Guidelines for diagnosis, treatment and follow-up. Annals of Oncology, 30 (8), 1194–1220. https://doi.org/10.1093/annonc/mdz173
- Liashchynskyi, P. B., Berezsky, O. M. (2024). Computer diagnostic systems: methods and tools. Ukrainian Journal of Information Technology, 6 (2), 57–63. https://doi.org/10.23939/ujit2024.02.057
- Berezsky, O. M., Liashchynskyi, P. B. (2024). Development of the architecture of a computer aided diagnosis system in medicine. Applied Aspects of Information Technology, 7 (4), 359–369. https://doi.org/10.15276/aait.07.2024.25
##submission.downloads##
Опубліковано
Як цитувати
Номер
Розділ
Ліцензія
Авторське право (c) 2025 Oleh Berezsky, Pavlo Liashchynskyi, Petro Liashchynskyi, Petro Selskyy

Ця робота ліцензується відповідно до Creative Commons Attribution 4.0 International License.
Закріплення та умови передачі авторських прав (ідентифікація авторства) здійснюється у Ліцензійному договорі. Зокрема, автори залишають за собою право на авторство свого рукопису та передають журналу право першої публікації цієї роботи на умовах ліцензії Creative Commons CC BY. При цьому вони мають право укладати самостійно додаткові угоди, що стосуються неексклюзивного поширення роботи у тому вигляді, в якому вона була опублікована цим журналом, але за умови збереження посилання на першу публікацію статті в цьому журналі.
Ліцензійний договір – це документ, в якому автор гарантує, що володіє усіма авторськими правами на твір (рукопис, статтю, тощо).
Автори, підписуючи Ліцензійний договір з ПП «ТЕХНОЛОГІЧНИЙ ЦЕНТР», мають усі права на подальше використання свого твору за умови посилання на наше видання, в якому твір опублікований. Відповідно до умов Ліцензійного договору, Видавець ПП «ТЕХНОЛОГІЧНИЙ ЦЕНТР» не забирає ваші авторські права та отримує від авторів дозвіл на використання та розповсюдження публікації через світові наукові ресурси (власні електронні ресурси, наукометричні бази даних, репозитарії, бібліотеки тощо).
За відсутності підписаного Ліцензійного договору або за відсутністю вказаних в цьому договорі ідентифікаторів, що дають змогу ідентифікувати особу автора, редакція не має права працювати з рукописом.
Важливо пам’ятати, що існує і інший тип угоди між авторами та видавцями – коли авторські права передаються від авторів до видавця. В такому разі автори втрачають права власності на свій твір та не можуть його використовувати в будь-який спосіб.










